·妇儿临床·
基于生物信息学途径探究卵巢癌关键预后基因
许梦莹 张广美
【摘要】 目的 应用生物信息学途径探究卵巢癌关键预后基因,有助于了解卵巢癌发生发展的分子机制,为卵巢癌患者提供新的治疗靶点。 方法 从基因表达数据库(gene expression omnibus,GEO)中下载浆液性卵巢癌上皮细胞和正常卵巢上皮细胞芯片数据GSE14407、GSE18520、GSE54388和GSE66957,用R语言软件筛选差异基因并得到共同差异基因。应用clusterProfiler软件对共同差异基因进行GO功能分析及KEGG通路富集分析。使用STRING建立蛋白互作用网络并使用cytoscape选出关键基因。采用survival包、survimner包对关键基因进行预后生存分析。 结果 经过筛选得到305个共同差异基因,其中250个基因表达上调,55个基因表达下调。共同差异基因主要富集于染色体分离、细胞周期G1/S转变、细胞黏附、细胞间连接、磷脂酰肌醇-3-激酶/丝苏氨酸蛋白激酶(PI3K-AKT)信号通路、Rap1信号通路、细胞增殖调控和粘附斑激酶信号通路等。蛋白互作用网络筛选得到14个关键基因,生存分析显示UBE2C基因的高表达导致卵巢癌患者的总生存率明显降低。 结论 UBE2C基因的表达与卵巢癌患者的总生存率密切相关,有望成为提高卵巢癌患者预后的新的生物学靶点。
【关键词】 卵巢癌; 基因表达数据库; 差异表达基因; 关键基因
作者单位:150001,哈尔滨医科大学附属第一医院妇产科
通讯作者:张广美(guangmeizhang@126.com)
Exploring key prognostic genes of ovarian cancer based on bioinformatics approach
XU Mengying, ZHANG Guangmei.
Department of Obstetrics and Gynecology, first affiliated Hospital of Harbin Medical University, Harbin Heilongjiang 150001, China
[Abstract] Objective The application of bioinformatics approach to explore the key prognostic genes of ovarian cancer is expected to understand the molecular mechanism of the occurrence and development of ovarian cancer, and provide a new therapeutic target for patients with ovarian cancer. Methods Microarray data of tumor ovarian and normal ovarian epithelial cells GSE14407, GSE18520, GSE54388 and GSE66957 were downloaded from Gene Expression Omnibus (GEO) database. We used R language to screen the differentially expressed genes and obtain the common differentially expressed genes. ClusterProfiler software was used for GO function analysis and KEGG pathway enrichment analysis of differential genes. Protein interaction network was constructed with STRING and key genes were screened by cytoscape. Survival package and survimner package were used for survival analysis of key genes. Results Total 305 DEGs (differentially expressed genes), including 250 up-regulated genes and 55 down-regulated genes, were screened in this study. DEGs were mainly enriched in chromosome segregation, G1/S transformation of cell cycle, cell adhesion, intercellular connectivity, PI3K-AKT signaling pathway, Rap1 signaling pathway, cell proliferation regulation and focal adhesion kinase signaling pathway.14 key genes were screened by protein interaction network, and survival analysis showed that the high expression of UBE2C gene led to a significant reduction in the overall survival rate of ovarian cancer patients. Conclusion The expression of UBE2C gene is closely related to the survival rate of ovarian cancer patients and is expected to become a new biological target to enhance the prognosis of ovarian cancer patients.
[Key words] Ovarian cancer; GEO; Differentially expressed genes; Key genes
卵巢癌的病死率居妇科肿瘤首位[1],因其发病隐匿,缺乏有效的早期诊断方法,超过70%的患者就诊时已为晚期[2-3],并且大多数已经发生了远处转移。大多数患者在2年内经历疾病复发,且复发性卵巢癌缺乏有效的治疗方案。随着DNA和RNA测序、DNA微阵列、高通量蛋白质组学和代谢组学等技术的日益普及,需要新的方法将这些新类型的数据转化为新信息,因此,通过高通量的基因芯片技术和生物信息学寻找卵巢癌有效的肿瘤标志物、关键预后基因,探索卵巢癌发生发展的机制具有重要意义,为卵巢癌患者的靶向治疗提供契机。考虑到基因芯片结果假阳性的可能,本研究综合几个基因芯片数据进行了分析。
资料与方法
一、基因芯片数据采集
从基因表达数据库GEO(www.ncbi.nlm.nih.gov/geo)中下载得到ID号为GSE14407、GSE18520[4]、GSE66957及GSE54388的浆液性卵巢癌及正常卵巢细胞mRNA芯片数据。其中GSE14407包含12例正常卵巢表面上皮细胞和12例浆液性卵巢癌上皮细胞,GSE18520包含10例正常卵巢表面上皮细胞和53例浆液性卵巢癌上皮细胞,GSE66957包含12例正常卵巢表面上皮细胞和57例浆液性卵巢癌上皮细胞,GSE54388包含6例正常卵巢表面上皮细胞和16例浆液性卵巢癌上皮细胞。
二、研究方法
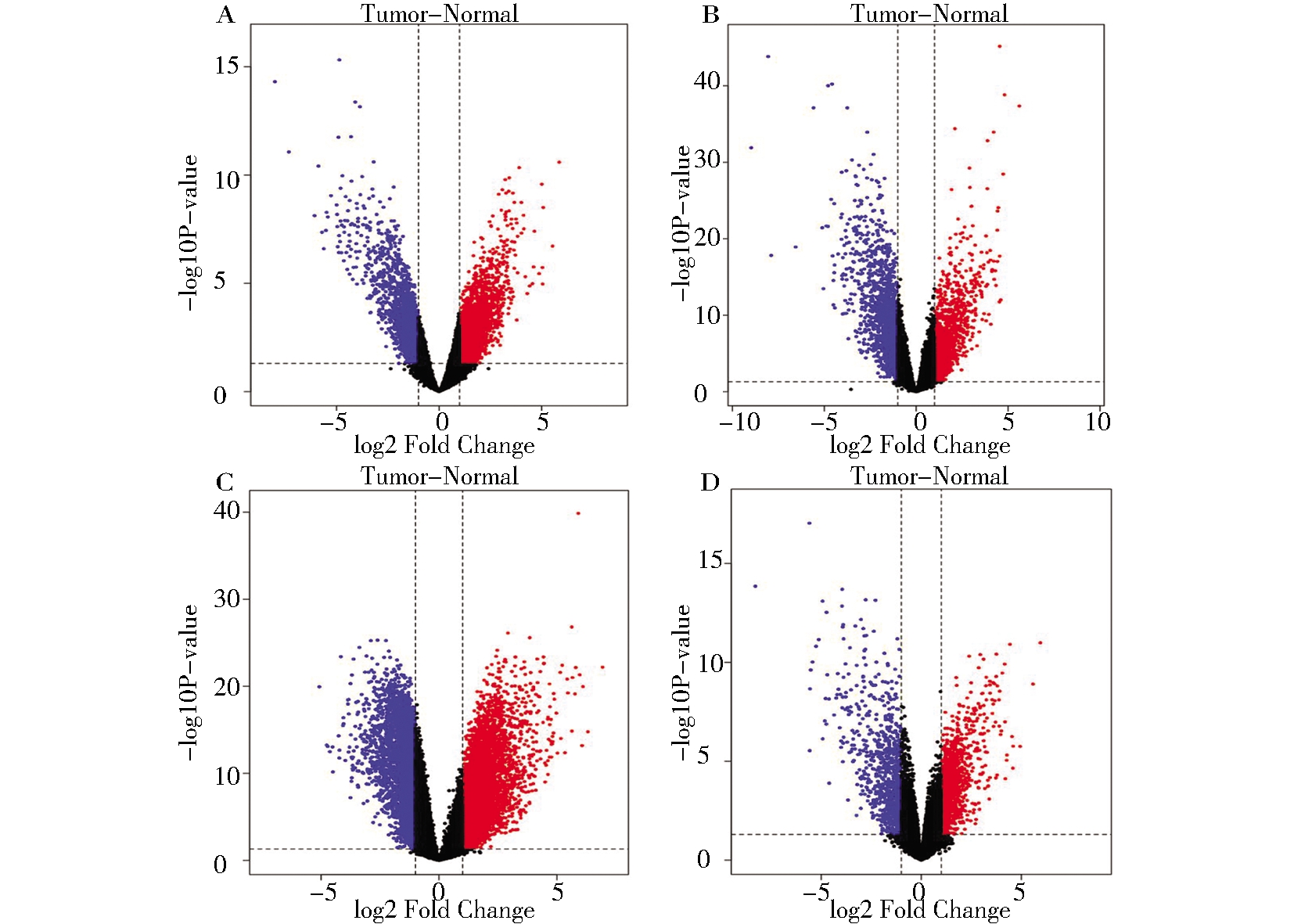
1. 共同差异基因的筛选:将GSE14407、GSE18520、GSE54388和GSE66957芯片数据导入R语言(https://www.r-project.org/)软件中,使用affy包对上述数据集的原始数据进行标准化处理,并运用Limma包(http://www.bioconductor.org/package/release/bioc/html/limma.html/)对GEO芯片数据进行差异计算,筛选阈值设为校正后P<0.05,差异表达倍数|logFC|≥2[5],并使用ggplot 2绘制各数据集差异基因的火山图,运用R VennDiagram对四个数据集中表达均为上调或下调的差异基因取交集得到共同差异基因,运用R heatmap对表达上调和下调的共同差异基因绘制热图。以下的生物信息学分析对象均为共同差异基因。
2.共同差异基因的功能富集:运用R clusterProfiler包(http://www.bioconductor.org/package/release/bioc/html/clusterProfiler.html/)对共同差异基因进行GO功能及KEGG通路富集[6](以P<0.05作为显著性富集的阈值)。
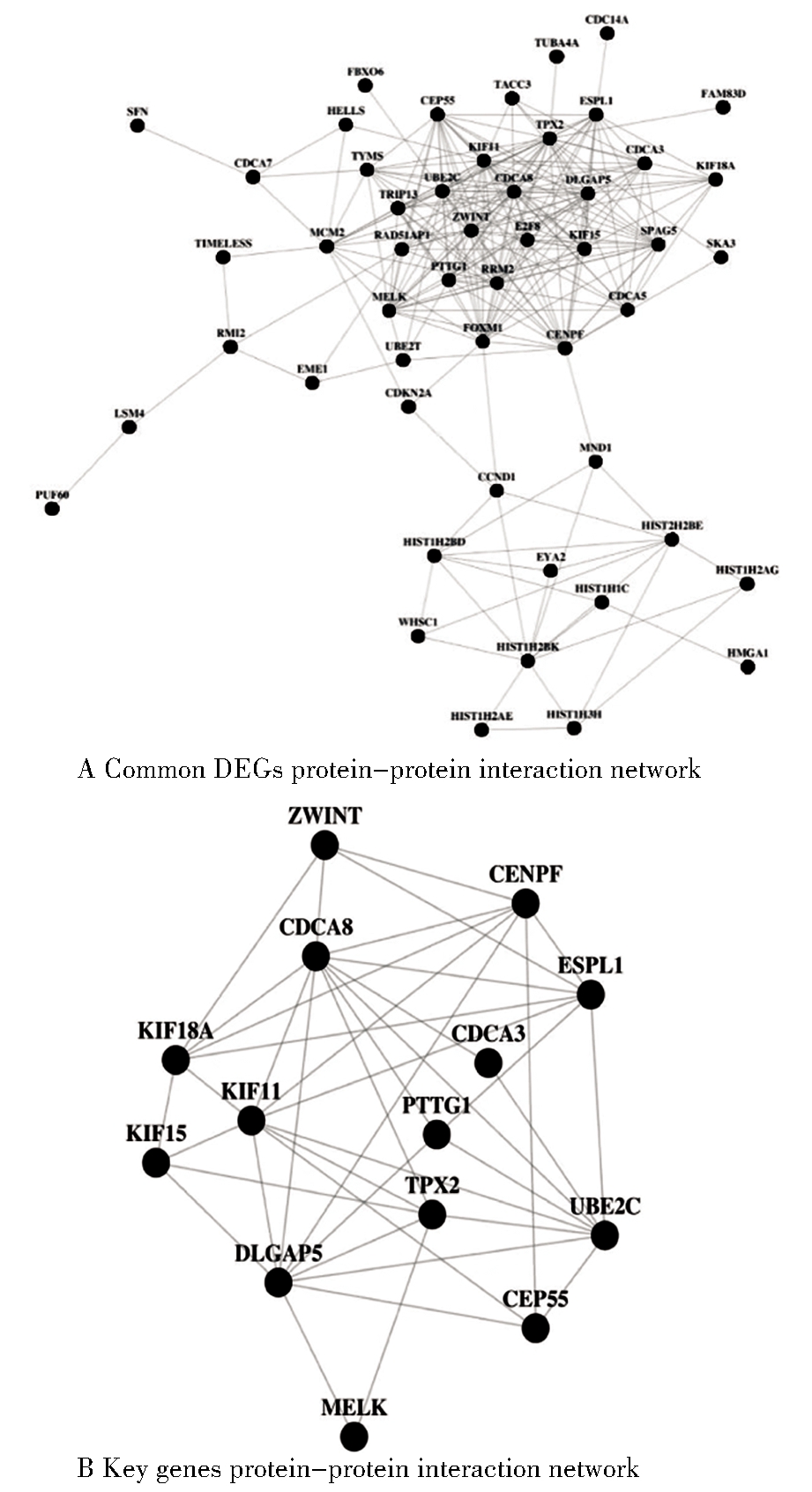
3. 建立蛋白质互作用网络:运用STRING(https://string-db.org)构建共同差异基因的蛋白相互作用(protein-protein interaction,PPI)网络[7],将最低互作用分值设置成高度可信(high confidence=0.8)[8],并将使用Cytoscape软件进行可视化分析,选出节点较多的关键基因并构建PPI核心网络。
4. 统计方法:使用survival包、survminer包分析关键差异基因的表达对卵巢癌患者生存率(病人样本来自TCGA数据库,以表达中位数为界,分为高表达组和低表达组)的影响,并用survminer包绘制生存曲线,通过Log-rank检验法比较生存曲线的差异,设置参数P<0.05为差异有统计学意义。
结 果
一、筛选出的共同差异基因
在GSE14407筛选得到共同差异基因6 572个,其中上调基因4 953个、下调基因1 619个;在GSE18520筛选得到共同差异基因3 648个,其中上调基因2 012个、下调基因1 636个;在GSE66957筛选得到共同差异基因9 342个,其中上调基因5 814个、下调基因3 528个;在GSE54388筛选得到共同差异基因2 435个,其中上调基因1 512个、下调基因923个(校正后P<0.05,差异表达倍数|logFC|≥2)。各数据集差异基因火山图见图1。四个数据集共筛选得到305个差异基因,其中250个表达上调的共同差异基因和55个表达下调的共同差异基因,韦恩图及热图见图2。
二、共同差异基因富集分析
富集分析结果(P<0.05)显示,在卵巢癌中表达上调的共同差异基因主要富集于染色体分离、细胞周期G1/S转变、细胞黏附、细胞间连接、磷脂酰肌醇-3-激酶/丝苏氨酸蛋白激酶(PI3K-AKT)信号通路和Rap1信号通路等;表达下调的共同差异基因则主要富集于细胞增殖调控、粘附斑激酶信号通路等,见表1及图3、图4。
表1 共同差异基因的GO功能和KEGG通路
Table 1 GO function and KEGG pathway analysis of common DEGs

ID 富集分析描述基因数(n)表达上调共同差异基因GO:0007059*染色体分离19GO:0048285*细胞器裂变19GO:0044843*细胞周期G1/S转变11GO:0098742*细胞黏附11GO:0005911*细胞间连接14KEGG:hsa04151*PI3K/AKT信号通路10KEGG:hsa04015*Rap1信号通路7KEGG:hsa04512*细胞外基质受体相互作用6表达下调共同差异基因GO:0050678*细胞增殖调控5
注:*P<0.05
三、蛋白质相互作用网络分析
蛋白互作用分析,将最低互作用分值设置成高度可信(high confidence=0.8)[8],得到包含51个节点的PPI网络。其中,PPI核心网络由相互作用关系较高的14关键基因构成,分别为ZWINT、CENPF、CDCA8、KIF18A、KIF15、KIF11、CDCA3、ESPL1、TPX2、DLGAP5、PTTG1、UBE2C、CEP55、MELK,提示上述基因在卵巢癌中起关键作用,见图5。
四、生存分析
分析ZWINT、CENPF、CDCA8、KIF18A、KIF15、KIF11、CDCA3、ESPL1、TPX2、DLGAP5、PTTG1、UBE2C、CEP55、MELK关键基因的表达水平与卵巢癌患者生存率之间的关系。其中仅UBE2C的表达高低与卵巢癌患者的生存率呈负相关。即UBE2C高表达患者的生存率明显低于该基因低表达患者,见图6。
讨 论
卵巢癌是女性生殖系统最致命的恶性肿瘤,是导致女性癌症患者死亡的第五大原因[1]。此外,其高转移率和化疗耐药引起的疾病复发是卵巢癌死亡的主要原因。因此,寻找可靠的肿瘤标志物,探索卵巢癌的分子机制对卵巢癌的诊断、治疗和预后具有重要意义。在本研究中,使用生物信息学方法整合了四个基因芯片数据集:GSE14407、GSE18520、GSE54388和GSE66957,使用R语言软件中的软件包筛选共得到305个共同差异基因,其中250个表达上调基因,55个表达下调基因。GO与KEGG分析显示共同差异基因主要富集于染色体分离、细胞周期G1/S转变、细胞黏附、细胞间连接、磷脂酰肌醇-3-激酶/丝苏氨酸蛋白激酶(PI3K-AKT)信号通路、Rap1信号通路、细胞增殖调控和粘附斑激酶信号通路等。
本研究通过在线数据库STRING对共同差异基因进行蛋白质相互作用分析,筛选得到节点较多的14个关键基因,对关键基因进行分析,其中仅泛素结合酶E2C(UBE2C)的高表达与卵巢癌患者的生存率呈负相关。UBE2C是泛素-蛋白酶体系统(ubiquitin-proteasome system,UPS)中泛素偶联酶E2家族的主要成员之一,泛素化酶主要分为3类:泛素激活酶(E1),负责激活泛素分子;泛素连接酶(E3),负责与靶蛋白结合;泛素结合酶(E2),在E3酶的介导下将泛素传给靶分子[9]。1997年Townsley等人初次克隆了人类的UBE2C基因[10],该基因位点位于20q13.12,编码的泛素结合酶由179个氨基酸组成,分子量为19.6KD[11],是人类细胞中被识别的第十个泛素结合酶基因,可通过激活有丝分裂后期促进复合物形成[12],通过参与泛素依赖的蛋白水解过程在细胞周期、信号转导、细胞分化[13]等多种正常细胞过程中发挥重要作用。UPS介导的泛素酶解过程依赖于其成员泛素活化酶E1、泛素结合酶E2、泛素连接酶E3酶链的顺序激活,UBE2C与经E1活化的泛素形成中间复合物,与E3相识别并在E3的催化作用下把泛素呈递给底物蛋白,如此反复最终将多泛素化的底物蛋白(如cyclinsA和cyclinsB等细胞周期相关蛋白)呈递给26S蛋白酶体并迅速分解[14],使得细胞进入正常有丝分裂中,通过终止纺锤体检查点信号使得细胞由中期进入后期,直至染色体正常分离,细胞完成正常有丝分裂。然而UBE2C异常高表达,使细胞染色体不稳定性增加,使得细胞逃离纺锤体检查点的监控作用,染色体不能正常分离或延迟分离而发生染色体紊乱,最终使得细胞不能完成正常分裂,形成非整倍体,这也是细胞癌变的特点之一[15]。Dai认为染色体延迟分离是UBE2C过表达导致染色体紊乱的主要原因[16]。Okamoto等通过实验研究表明UBE2C在肺癌、结肠癌、乳腺癌、胰腺癌、骨肉瘤等多种肿瘤中高表达,并促进细胞的增殖和恶变从而发生致癌基因的作用[17],尤其是恶性程度高、分化程度低、转移倾向高的癌症,往往表现为UBE2C高表达、患者低生存率[18]。Martinez-canales基于生物信息学在卵巢癌中的研究发现,在6%的卵巢癌组织中发现了UBE2C基因的上调和扩增,是具有治疗干预潜力的基因[19]。
本研究应用生物信息学方法综合分析了浆液性卵巢癌共同差异基因,结果得到了关键基因UBE2C,其与卵巢癌患者生存率成负相关,即UBE2C高表达患者的生存率明显低于低表达患者,提示其可能是改善卵巢癌患者预后的生物学靶点,但在卵巢癌中的分子机制尚不明确,需进一步研究。
参考文献
1 Siegel RL,Miller KD and Jemal A.Cancer statistics,2018. CA Cancer J Clin,2018,68:7-30.
2 Hu H,Huang G,Wang H,et al.Inhibition effect of triptolide on human epithelial ovarian cancer via adjusting cellular immunity and angiogenesis.Oncol Rep,2018,39:1191-1196.
3 Rice MS,Rist PM,Winter AC,et al.Migraine and invasive epithelial ovarian cancer risk in the Nurses′ Health Study II and the Women′s Health Study.Int J Cancer,2018,142:534-539.
4 Liu J,Li S,Liang J,et al.ITLNI identified by comprehensive bioinformatic analysis as a hub candidate biological target in human epithelial ovarian cancer.Cancer Manag Res,2019,11:2379-2392.
5 Yang X,Zhu SM,Li L,et al.Identification of differentially expressed genes and signaling pathways in ovarian cancer by integrated bioinformatics analysis.Onco Targets Ther,2018,11:1457-1474.
6 Zhou Y,Layton O,Hong L,et al.Identification of Genes and Pathways Involved in Ovarian Epithelial Cancer by Bioinformatics Analysis.J Cancer,2018,9:3016-3022.
7 Xue J,Yang G,Ding H,et al.Role of NSC319726 in ovarian cancer based on the bioinformatics analyses.Onco Targets Ther,2015,8:3757-3765.
8 王旦旦,吕琳,陈智如,等.顺铂耐药相关基因的筛选、验证及生物信息学分析.重庆医学,2019,48:124-128.
9 王少冰,王芳.泛素结合酶E2C与肿瘤的关系研究进展.医学研究生学报,2015,28:543-548.
10 Townsley FM,Aristarkhov A,Beck S,et al.Dominant-negative cyclin-selective ubiquitin carrier protein E2-C/UbcH10 blocks cells in metaphase.Proc Natl Acad Sci U S A,1997,94:2362-2367.
11 Zhang HQ,Zhao G,Ke B,et al.Overexpression of UBE2C correlates with poor prognosis in gastric cancer patients.Eur Rev Med Pharmacol Sci,2018,22:1665-1671.
12 韩少山,孙昊,刘青光.UbcH10与肿瘤关系研究进展.中国肿瘤临床,2011,38:867-869.
13 Williamson A,Wickliffe KE,Mellone BG,et al.Identification of a physiological E2 module for the human anaphase-promoting complex.Proc Natl Acad Sci USA,2009,106:18213-18218.
14 Wagner KW,Sapinoso LM,El-Rifai W,et al.Overexpression,genomic amplification and therapeutic potential of inhibiting the UbcH10 ubiquitin conjugase in human carcinomas of diverse anatomic origin.Oncogene,2004,23:6621-6629.
15 van Ree JH,Jeganathan KB,Malureanu L,et al.Overexpression of the E2 ubiquitin-conjugating enzyme UbcH10 causes chromosome missegregation and tumor formation.J Cell Biol,2010,188:83-100.
16 Dai W,Wang Q,Liu T,et al.Slippage of mitotic arrest and enhanced tumor development in mice with BubR1 haploinsufficiency.Cancer Res,2004,64:440-445.
17 Okamoto Y,Ozaki T,Miyazaki K,et al.UbcH10 is the cancer-related E2 ubiquitin-conjugating enzyme.Cancer Res,2003,63:4167-4173.
18 Cacciola NA,Calabrese C,Malapelle U,et al.UbcH10 expression can predict prognosis and sensitivity to the antineoplastic treatment for colorectal cancer patients.Mol Carcinog,2016,55:793-807.
19 Martinez-Canales S,Lopez de Rodas M,Nuncia-Cantarero M,et al.Functional transcriptomic annotation and protein-protein interaction analysis identify EZH2 and UBE2C as key upregulated proteins in ovarian cancer.Cancer Med,2018,7:1896-1907.
(收稿日期:2019-11-14)