·遗传与出生缺陷·
中国西北地区苯丙氨酸羟化酶基因突变构成分析
何江 强荣 毛新梅 徐发亮 闫有圣 余伍忠 史清海
基金项目:新疆维吾尔自治区自然科学基金面上项目(2016D01C393);国家自然科学基金面上项目(81871020).
作者单位:830000 乌鲁木齐,新疆军区总医院检验科(何江,余伍忠,史清海);陕西省妇幼保健院医学遗传中心(强荣);宁夏回族自治区妇幼保健院新生儿疾病筛查中心(毛新梅);青海省妇幼保健院新生儿疾病筛查中心(徐发亮);甘肃省妇幼保健院医学遗传中心(闫有圣)
通讯作者:史清海(shiqinghai@aliyun.com)
【摘要】 目的 分析中国西北地区苯丙氨酸羟化酶(PAH)基因的突变特征,为苯丙酮尿症(PKU)基因诊断及遗传咨询提供理论依据。方法联合应用PCR产物直接测序法、基因芯片捕获和二代高通量测序技术对2003年1月—2019年12月在中国西北五省区新生儿疾病筛查中心确诊的326例PKU患儿及其父母的PAH基因启动子、第1~13外显子及其旁侧内含子区域进行基因突变分析。结果在中国西北地区326例PKU患者652条PAH等位基因中检测出105种致病突变,总检出率为87.7%,突变以错义突变、剪切位点突变、无义突变和移码突变为主;大部分突变主要分布在E7(25.2%)、E6(11.8%)、E3(9.2%)、E12(9.2%)、E11(8.3%)、I4(5.2%)、E2(3.5%)和E5(3.5%)中;突变频率较高的PAH基因致病突变是R243Q,与中国北方、韩国比较一致,但显著区别于日本(R413P)、德国(R408W)、以色列(IVS10-11G>A)、意大利(R261Q)、巴西(V388M)和美国(R408W)等国家,其他常见突变类型还包括EX6-96A>G、IVS4-1G>A、R413P、R111X、Y356X、R53H、R241C和IVS7+2T>A;中国西北汉族与西北维吾尔族、回族相比,R243Q、R413P和EX6-96A>G突变的检出率具有显著性差异(P<0.05)。中国西北地区R243Q突变检测率显著高于日本、韩国(P<0.05)。国际上首次报道14种新突变,在中国人群中首次报道9种突变。结论中国西北地区PKU患者PAH基因突变构成与中国北方基本一致,稍区别于日本、韩国等东亚国家,但显著不同于其他西亚、欧洲、美洲国家。中国西北地区不同民族PAH基因突变谱具有其独立保守的特性。中国R243Q突变的起源可能在中国汉族中,R241C、Q232X突变频率的高处在中国回族中,而R413P、F161S、IVS10-11G>A、P281L和IVS11-2A>C突变频率的高位在中国维吾尔族中。
【关键词】 苯丙酮尿症; 苯丙氨酸羟化酶; 突变; 基因测序
Analysis on mutation spectrum of phenylalanine hydroxylase gene in Northwest Chinese patients with phenylketonuria
HE Jiang, QIANG Rong, MAO Xinmei,XU Faliang, YAN Yousheng, YU Wuzhong, SHI Qinghai.
Department of Laboratory Medicine, General Hospital of Xinjiang Military Region, PLA,Urumqi 830000, China
[Abstract] Objective To investigate the mutation characteristics of the phenylalanine hydroxylase(PAH)gene in Northewest Chinese patients with phenylketonuria(PKU)and to provide a theoretical basis for genetic diagnosis and consultation of PKU at the prenatal stage.MethodsThrough combining the methods of PCR direct sequencing, gene chip and high throughput sequencing, we detected the mutations in PAH gene promoter, 1-13 exons and adjacent introns among 326 children and their patients, who were recruited from the patients diagnosed by the neonatal disease screening Centers in the five northwestern provinces of China from January 2003 to December 2019.ResultsAmong 326 PKU patients, out of 652 alleles, 105 different mutations were detected. The total detection rates were 87.7%. The major types of mutations are missense mutation, splicing mutation, nonsense mutation and frameshift mutations. Most mutations are located in exon 79 (25.2%), followed by exon 6 (11.8%), exon 3 (9.2%), exon 12 (9.2%), exon 11 (8.3%), intron 4 (5.2%), exon 2 (3.5%) and exon 2 (3.5%). The most common mutation was R243Q, which is the same to the most common mutation in Northern China and Korea, but different from that in Japan (R413P), Germany (R408W), Israel (IVS10-11G>A), Italy (R261Q), Brazil (V388M) and the United States (R408W). The other common mutations included EX6-96A>G, IVS4-1G>A, R413P, R111X, Y356X, R53H, R241C and IVS7+2T>A. The frequencies of R243Q, R413P and EX6-96 A>G mutations of ethnic Han group were significantly different from those of Uygur group. In Northwest China, the frequency of R243Q mutation was significantly higher than that in Japan or Korea (P<0.05). Fourteen new PAH gene mutations were reported for the first time. Additional nine mutations were first identified among the Chinese population.ConclusionAmong Northwestern Chinese, mutation spectrum of PAH gene is similar to that in Northern China, but slightly different from that in Japan, Korea and other East Asian countries, or significantly different from that in West Asian, European and American countries. The PAH gene profiles in Northwest China are independent and conservative. The R243Q mutation is probably originated from Han ethnicity; R241C and Q232X are from Hui ethnicity; and R413P, F161S, IVS10-11G>A, P281L and IVS11-2A>C are from Uygur ethnicity.
Key words] phenylketonuria; phenylalanine hydroxylase; mutation; gene sequencing
苯丙酮尿症(phenylketonuria,PKU)是一种由于苯丙氨酸羟化酶(phenylalanine hydroxylase,PAH)基因突变导致酶活性降低或丧失,导致苯丙氨酸(phenylatanine,Phe)代谢障碍的常染色体隐性遗传病。PKU在世界各国发病率差异较大,在中国西北地区发病率为1∶3 761,明显高于华北(1∶8 218)和东北地区(1∶7 084),也高于全国平均水平(1∶11 444)[1-2]。PAH基因突变的种类、频率和分布在不同国家、地区和民族间也存在较大差异,表现出极大的遗传异质性[3]。中国西北地区是古今“丝绸之路”连接中国内地与亚欧各国文化、贸易的大通道之一,不同国家、民族、肤色的人种沿此东西向通道流动迁徙、居住生活、融合通婚,对本地区的血缘融合和基因交流产生了广泛影响。对西北地区不同民族及地域的PKU患者进行PAH基因突变谱研究,对于指导本地区PKU患者的产前诊断、PAH基因携带者筛查及了解基因的进化、漂移具有重要科学价值。本文对西北地区确诊的326例PKU患者及其父母进行PAH基因突变分析,以探究西北地区PAH基因的突变规律及分布特征。
对象与方法
一、研究对象及诊断标准
选取2003年1月—2019年12月经中国西北五省区新生儿疾病筛查中心筛查发现或各医院儿科门诊遗传咨询发现并确诊的326例PKU患者为研究对象,其中男165例,女161例;新疆118例,甘肃29例,青海47例,宁夏70例, 陕西62例;汉族223例,回族67例,维吾尔族33例,哈萨克族1例,土族1例,藏族1例。年龄范围从2个月至16岁,平均年龄(1.5±0.7)岁。初诊血清Phe浓度为216~25 680 μmol/L。
高苯丙氨酸血症(HPA)确诊标准:对出生72 h后充分哺乳新生儿采集足跟血,对门诊患儿采集静脉血,测定Phe浓度>120 μmol/L者进行召回复查,复查Phe浓度仍然>120 μmol/L,首先确诊为HPA。
PKU诊断标准:复查Phe浓度>120 μmol/L,采集HPA患儿血样进行血二氢蝶呤还原酶(DHPR)测定,采集尿液标本进行尿蝶呤谱分析,进行四氢生物蝶呤(BH4)负荷试验或(>600 μmol/L)或者Phe和BH4联合负荷试验(≤600 μmol/L),如患者DHPR活性正常,BH4口服或者Phe+BH4联服4 h后血Phe浓度无明显变化,尿新蝶呤(neopterin,N)与生物蝶呤(biopterin,B)比值正常,BH4(+),经基因测序发现存在2个(极个别病例具有2个以上PAH基因致病突变)且突变经溯源分别来源于其生物学父母,考虑为经典型PKU。
对Phe>600 μmol/L患儿经确诊后立即行低苯丙氨酸饮食控制治疗。所有受试者均签署知情同意书。本研究经本院伦理委员会批准实施。
二、方法
1.主要试剂与仪器:TaqDNA聚合酶和dNTPs(Promega公司),引物(上海生工公司合成),QIAamp DNA Blood Midi Kit (Qiagen, Hilden, Germany);T4 DNA聚合酶、T4磷酸核苷酸激酶、Klenow 聚合酶、dATP和Klenow 3′-5′ exo-enzyme、NimbleGen SeqCap EZ (生物素化寡DNA核酸探针)和Agencourt AMPure SPRI beads(美国Illumina公司);饱和酚、氯仿、已戊醇(北京化学试剂公司),蛋白酶K(德国Merck公司),其他试剂自配。Covaris S2超声波仪(Massachusetts, USA),3130XL型基因测序仪(美国ABI公司),PCR扩增仪(PE公司9700型),恒温循环仪及垂直恒温电泳槽(北京君意东方电泳设备有限公司),三恒多用电泳仪(北京六一仪器厂)。
2. 标本采集及DNA提取:抽取患者及父母外周静脉血1 ml,滴于滤纸上呈3~5个滤纸斑,阴凉干燥避光处晾干,1~2 h后装入无菌干净密封袋内留存,剩余全血置4℃冰箱保存。血斑采用快速酚/氯仿抽提法,全血采用QIAamp DNA Blood Midi Kit提取法,提取的基因组DNA保存于-20℃备用。
3. PCR扩增及序列测定:PAH基因启动子及第1~13外显子的引物序列设计参照文献[4],在美国ABI 9700型扩增仪上进行PCR反应。PCR总体积20 μL, 包括DNA模板2 μL,25 μmol/L引物0.5 μL,2.5 mmol/L dNTP 4 μL,3 U/mL Taq DNA聚合酶1μL,1×PCR buffer 5 μL,dd H2O 7.5 μL。各成分混匀后先95℃预变性15 min,然后进入95℃变性45 s,62℃复性45 s,72℃延伸60 s,循环11次;再95℃变性45 s,57℃复性45 s,72℃延伸60 s,循环24次;最后72℃ 7min后降温至4℃备用。PCR产物用2%琼脂糖凝胶电泳检测。详细操作步骤参照文献[5-6]。
258例患者及其父母在2013年12月前均采用PCR产物直接测序法,样品纯化及序列分析应用美国ABI 3130 XL型序列分析仪完成。每个测序样品进行两次PCR,分别测定正、反方向的基因序列,所有序列变异位点均针对该位点,检测患者父母的DNA序列,以确定序列变异来源。如有新突变同时对患者双亲进行序列分析予以溯源验证。
4.应用基因芯片捕获和二代高通量测序技术检测PAH基因突变:68例患者全血样本(含Sanger测序未检出)从2014年1月开始采用QIAamp DNA Blood Midi Kit 提取基因组DNA。质检合格的基因组DNA经打断成片段后纯化,进行末端修复、接头连接、纯化后,纯化过的DNA片段经过ligation-mediated PCR (LM-PCR)进行扩增和纯化,使用NimbleGen SeqCap EZ 人全外显子捕获系统对苯丙氨酸羟化酶及其辅酶合成及代谢相关的五个基因(PAH, PTS, QDPR, GCH1, PCBD1,共计9 343 bp)进行目标序列捕获,被捕获的片段随后进行LM-PCR反应,生成测序用的DNA库。DNA库在Illumina HiSeq2000测序平台上进行扩增并进行高通量测序,连续双向测序90个循环。每个样本的平均测序深度大于100×以保证信息分析要求。测序参数为目标区平均覆盖度为98.9%,平均深度(X)为250.1,平均深度>30%位点所占比例93.6%,每条测序序列产生90 bp的双向测序读长。使用Illumina basecalling 软件(版本1.7)进行原始图像数据处理,测序数据分析使用Illumina分析流程。如患者检测出已知或者未知基因突变位点,对患者父母进行该突变位点PCR扩增并进行Sanger法测序,以确定序列变异真实性及其来源。
5.突变命名和验证:已知突变的命名结合测序结果参照http://pahab.mcgill.ca提供的突变名称命名。新突变的命名参照http://www.hgvs.org /mutnomen提供的命名法来命名。新序列变异通过查阅PAH数据库(http://www.biopku.org/pah)、国际人类基因突变数据库Pubmed (http://www.ncbi. nlm.nih.gov/Pubmed)来确定,所有新变异均通过100名健康无关个体相应外显子测序排除为多态性位点后被认定为新突变。
6.统计学处理:各组计数资料以率(%)表示,采用SPSS 15.0软件进行数据统计和分析,组间比较采用χ2检验,以P<0.05为差异有统计学意义。
结 果
一、西北地区汉族PKU患者PAH基因突变分布及特点
在西北地区326例PKU患者652条PAH等位基因中共检出105种PAH基因致病突变,总检出率为87.7%,突变形式主要包括错义突变68种(64.8%)、剪切位点突变18种(17.1%),无义突变9种(8.6%),移码突变7种(6.7%),缺失突变3种(2.9%)。本研究在外显子区(除外显子13外)检测到500个外显子(exon,E)突变(76.7%),还在内含子(intron,I)2、内含子4、内含子7-8、和内含子10-12中检测到70个剪切位点突变(10.7%),在5′-UTR中检测到2个突变,PAH致病基因突变主要分布在E7(25.2%)、E6(11.8%)、E3(9.2%)、E12(9.2%)、E11(8.3%)、I4(5.2%)、E2(3.5%)和E5(3.5%),这八个区域所检出突变检出率占比为86.5%(495/572);突变频率较高的PAH基因致病突变是R243Q(22.1%)、EX6-96A>G(6.3%)、IVS4-1G>A(5.2%)、R413P(4.9%)、R111X(4.4%)、Y356X(3.7%)、R53H(3.4%)、R241C(2.8%)、IVS7+2T>A(2.0%),其余突变大多散在发生(<2.0%),47种突变只检测到1个等位基因,每种突变的发生率仅为0.2%。
表1 西北地区PKU患者PAH基因突变构成(%)
Table 1 Frequency of PAH genes mutations among the analyzed PKU patients in Northwest China (%)

Changes in amino acidsChanges in bases Mutation sitesMutation types Allele numbersRelative frequencies(%)5′-Flanking-626G>Ca-626G>C5′-terminalMissence10.25′-Flanking-480DelACTa-480DelACT5′-terminalDeletion10.2M1Iac.3G>TE1Missence10.2S16fsX38ac.47_48delCTE1Frameshift20.3A47Ec.140C>AE2Missence10.2R53Hc.158G>AE2Missence223.4IVS2+19T>Cc.168+19T>CI2Splicing10.2H64fsX9bc.190delCE3Frameshift10.2I65Tc.194T>CE3Missence91.4I65Sc.194T>GE3Missence20.3S70delc.208_210delTCTE3Deletion91.4R71Hc.212G>AE3Missence10.2T81Nbc.242C>AE3Missence10.2N93fsX5ac.279_282delCATCE3Frameshift20.3I95delc.284_286delTCAE3Deletion10.2A104Dc.311C>AE3Missence20.3H107Rac.320A>GE3Missence30.5R111Xc.331C>TE3Missence294.4P119Sc.355C>TE4Missence10.2P147Lc.440C>tE4Missence20.3IVS4-1G>Ac.442-1G>AI4Splicing345.2R155Hc.500G>AE5Missence20.3A156Pc.466G>CE5Missence10.2R158Wc.472C>TE5Missence10.2R158Qc.473G>AE5Missence40.6F161Sc.482T>CE5Missence101.5Y166Xc.498C>GE5Nonsence40.6R169Cbc.505C>TE5Missence10.2H170Qc.510T>AE6Missence40.6G171Eac.512G>AE6Missence10.2R176Xc.526C>TE6Nonsence40.6E182Kc.544G>AE6Missence10.2G188D/Vc.563G>A/TE6Missence20.3S196fsX4c.584-585insAE6Frameshift10.2T200fsX6c.598_599insAE6Frameshift10.2EX6-96A>Gc.611A>GE6Splicing416.3D222Gac.665A>GE6Missence20.3N223Iac.668A>TE6Missence10.2I224Tc.671T>CE6Missence20.3P225Sac.673C>TE6Missence10.2V230Ic.688G>AE6Missence50.8V230Abc.689T>CE6Missence10.2Q232Xc.694C>TE6Nonsence101.5R241Cc.721C>TE7Missence182.8R241Hc.722G>AE7Missence30.5R241fsX5c.722delGE7Frameshift20.3L242Fc.724C>TE7Missence10.2R243Xc.727C>TE7Nonsence10.2R243Qc.728G>AE7Missence12321.9G247Rc.739G>CE7Missence10.2G247Vc.740G>TE7Missence30.5R252Qc.755G>AE7Missence10.2
表1(续)
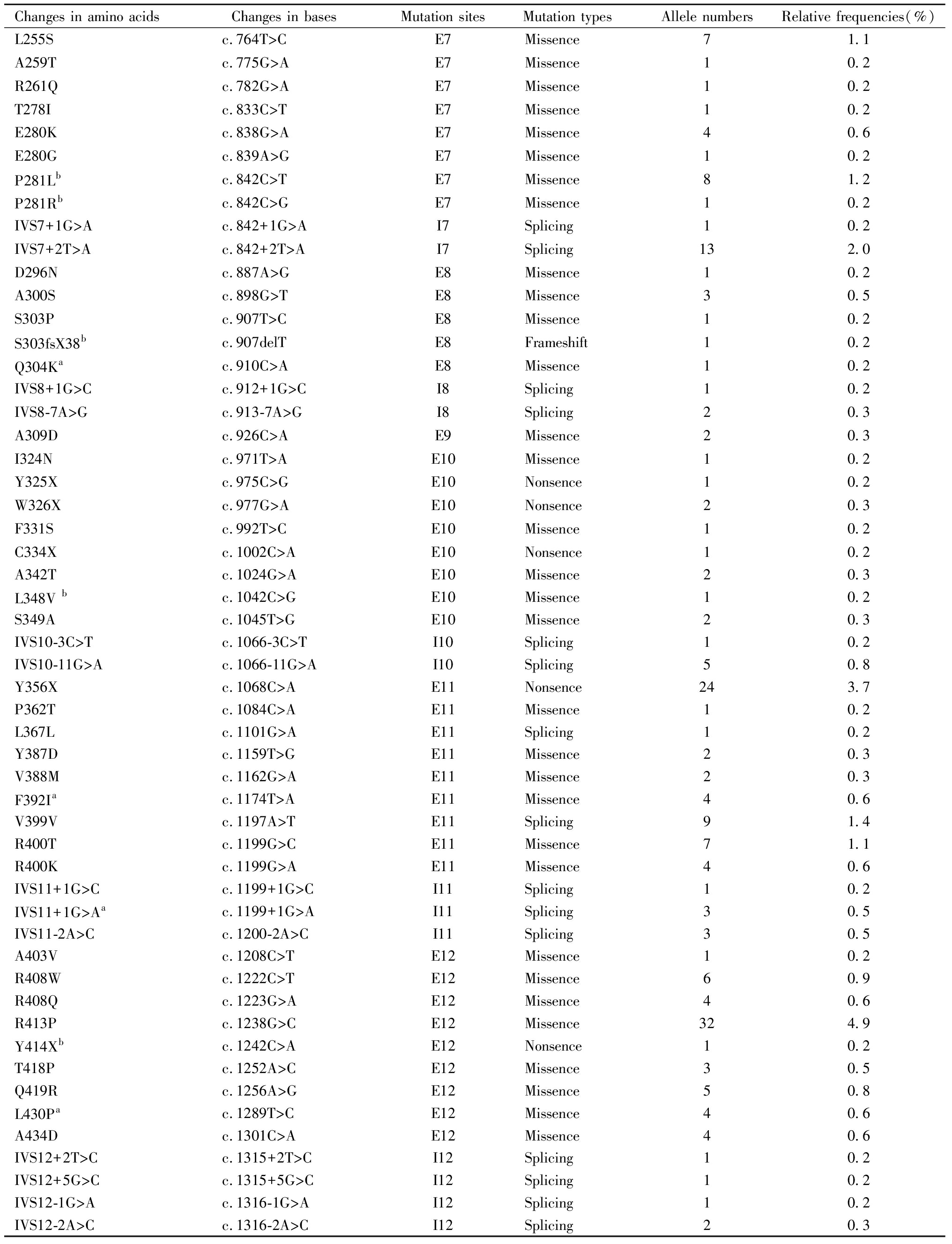
Changes in amino acidsChanges in bases Mutation sitesMutation types Allele numbersRelative frequencies(%)L255Sc.764T>CE7Missence71.1A259Tc.775G>AE7Missence10.2R261Qc.782G>AE7Missence10.2T278Ic.833C>TE7Missence10.2E280Kc.838G>AE7Missence40.6E280Gc.839A>GE7Missence10.2P281Lbc.842C>TE7Missence81.2P281Rbc.842C>GE7Missence10.2IVS7+1G>Ac.842+1G>AI7Splicing10.2IVS7+2T>Ac.842+2T>AI7Splicing132.0D296Nc.887A>GE8Missence10.2A300Sc.898G>TE8Missence30.5S303Pc.907T>CE8Missence10.2S303fsX38bc.907delTE8Frameshift10.2Q304Kac.910C>AE8Missence10.2IVS8+1G>Cc.912+1G>CI8Splicing10.2IVS8-7A>Gc.913-7A>GI8Splicing20.3A309Dc.926C>AE9Missence20.3I324Nc.971T>AE10Missence10.2Y325Xc.975C>GE10Nonsence10.2W326Xc.977G>AE10Nonsence20.3F331Sc.992T>CE10Missence10.2C334Xc.1002C>AE10Nonsence10.2A342Tc.1024G>AE10Missence20.3L348V bc.1042C>GE10Missence10.2S349Ac.1045T>GE10Missence20.3IVS10-3C>Tc.1066-3C>TI10Splicing10.2IVS10-11G>Ac.1066-11G>AI10Splicing50.8Y356Xc.1068C>AE11Nonsence243.7P362Tc.1084C>AE11Missence10.2L367Lc.1101G>AE11Splicing10.2Y387Dc.1159T>GE11Missence20.3V388Mc.1162G>AE11Missence20.3F392Iac.1174T>AE11Missence40.6V399Vc.1197A>TE11Splicing91.4R400Tc.1199G>CE11Missence71.1R400Kc.1199G>AE11Missence40.6IVS11+1G>Cc.1199+1G>CI11Splicing10.2IVS11+1G>Aac.1199+1G>AI11Splicing30.5IVS11-2A>Cc.1200-2A>CI11Splicing30.5A403Vc.1208C>TE12Missence10.2R408Wc.1222C>TE12Missence60.9R408Qc.1223G>AE12Missence40.6R413Pc.1238G>CE12Missence324.9Y414Xbc.1242C>AE12Nonsence10.2T418Pc.1252A>CE12Missence30.5Q419Rc.1256A>GE12Missence50.8L430Pac.1289T>CE12Missence40.6A434Dc.1301C>AE12Missence40.6IVS12+2T>Cc.1315+2T>CI12Splicing10.2IVS12+5G>Cc.1315+5G>CI12Splicing10.2IVS12-1G>Ac.1316-1G>AI12Splicing10.2IVS12-2A>Cc.1316-2A>CI12Splicing20.3
Note:a new mutations first reported by this study; b mutations which have been reported in other populations than Chinese by previous studies and are detected by this study.
二、中国西北地区汉族、维吾尔族和回族PAH基因常见致病突变检出率比较
R243Q是西北汉族和回族PAH基因最常见的突变类型,不同于西北维吾尔族突变检出率最高为R413P(12.1%);R243Q在西北汉族PKU患者中的的检出率显著高于西北回族和维吾尔族(P<0.05),R413P突变在西北维吾尔族中的检出率显著高于西北汉族和回族(P<0.05)。EX6-96 A>G突变在西北回族中的检出率显著低于西北汉族和维吾尔族(P<0.05)。西北地区R243Q突变频率的高处在汉族中,西北地区R241C、Q232X突变频率的高处在回族中,R413P、F161S、IVS10-11G>A、P281L和IVS11-2A>C突变的高处在维吾尔族中。
表2 中国西北地区汉族、维吾尔族和
回族PAH基因常见致病突变检出率比较(%)
Table 2 Detection rate of common mutation
in PAH genes by Han, Uygur or Hui ethnic group,
in Northwest China (%)
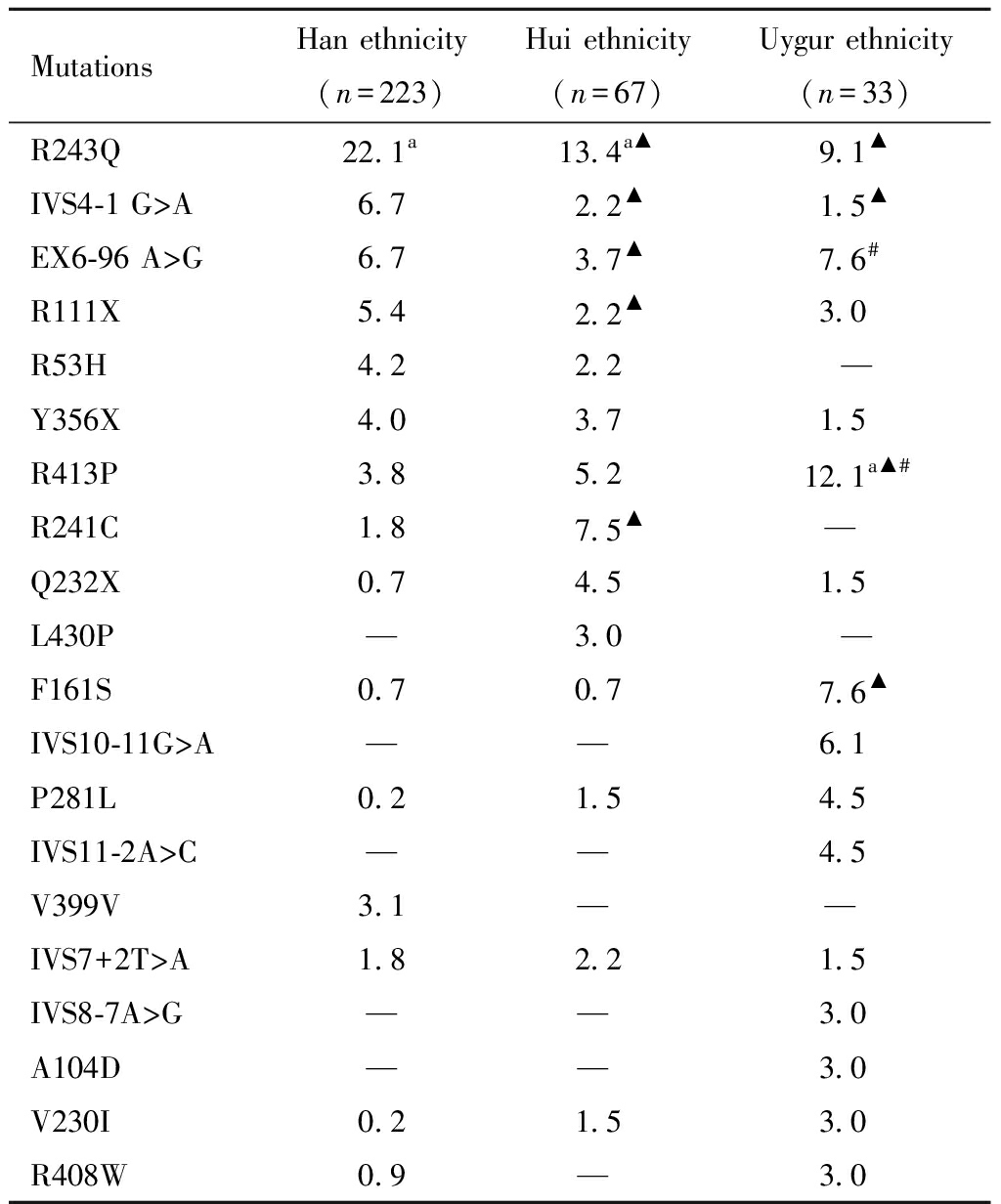
MutationsHan ethnicity(n=223)Hui ethnicity(n=67)Uygur ethnicity(n=33)R243Q22.1a13.4a▲9.1▲IVS4-1 G>A6.72.2▲1.5▲EX6-96 A>G6.73.7▲7.6#R111X5.42.2▲3.0R53H4.22.2—Y356X4.03.71.5R413P3.85.212.1a▲#R241C1.87.5▲— Q232X0.74.51.5L430P—3.0—F161S0.70.77.6▲IVS10-11G>A—— 6.1P281L0.21.54.5IVS11-2A>C—— 4.5V399V3.1— — IVS7+2T>A1.82.21.5IVS8-7A>G—— 3.0A104D—— 3.0V230I0.21.53.0R408W0.9— 3.0
Note:a mutation with the highest detection rate. - mutations that are not detected in this study. ▲ The mutation frequency in Uygur group is significantly different from that in Han group (P<0.05). # The mutation frequency in Uygur group is significantly different from that in Hui group (P<0.05).
三、中国西北地区PAH基因常见致病突变检出率与其他国家和地区的比较
如表3所示,中国西北地区突变频率较高的PAH基因致病突变是R243Q,与中国北方、韩国比较一致,但显著区别于日本(R413P)、德国(R408W)、以色列(IVS10-11G>A)、意大利(R261Q)、巴西(V388M)和美国(R408W)等国家。中国西北和中国北方地区的R243Q突变检出率显著高于日本、韩国(P<0.05),EX6-96A>G突变检出率显著低于中国北方、韩国(P<0.05),R111X和Y356XPAH基因突变检出率显著低于中国北方(P<0.05)。R243Q、R413P、IVS4-1 G>A和EX6-96A>G等在中国西北、中国北方、日本、韩国等东亚地区检出率较高的常见突变类型,在伊朗、以色列、德国、意大利、美国、巴西等其他西亚、欧洲及南北美洲国家却极为罕见。中国西北PAH基因突变分布与中国北方地区与中国北方基本一致,稍区别于日本、韩国等东亚国家,但显著不同于其他亚洲、欧洲、美洲国家。
表3 中国西北地区PAH基因常见突变频率与其他国家和地区的比较(%)
Table 3 Frequency of common mutation in PAH genes by regions or countries(%)
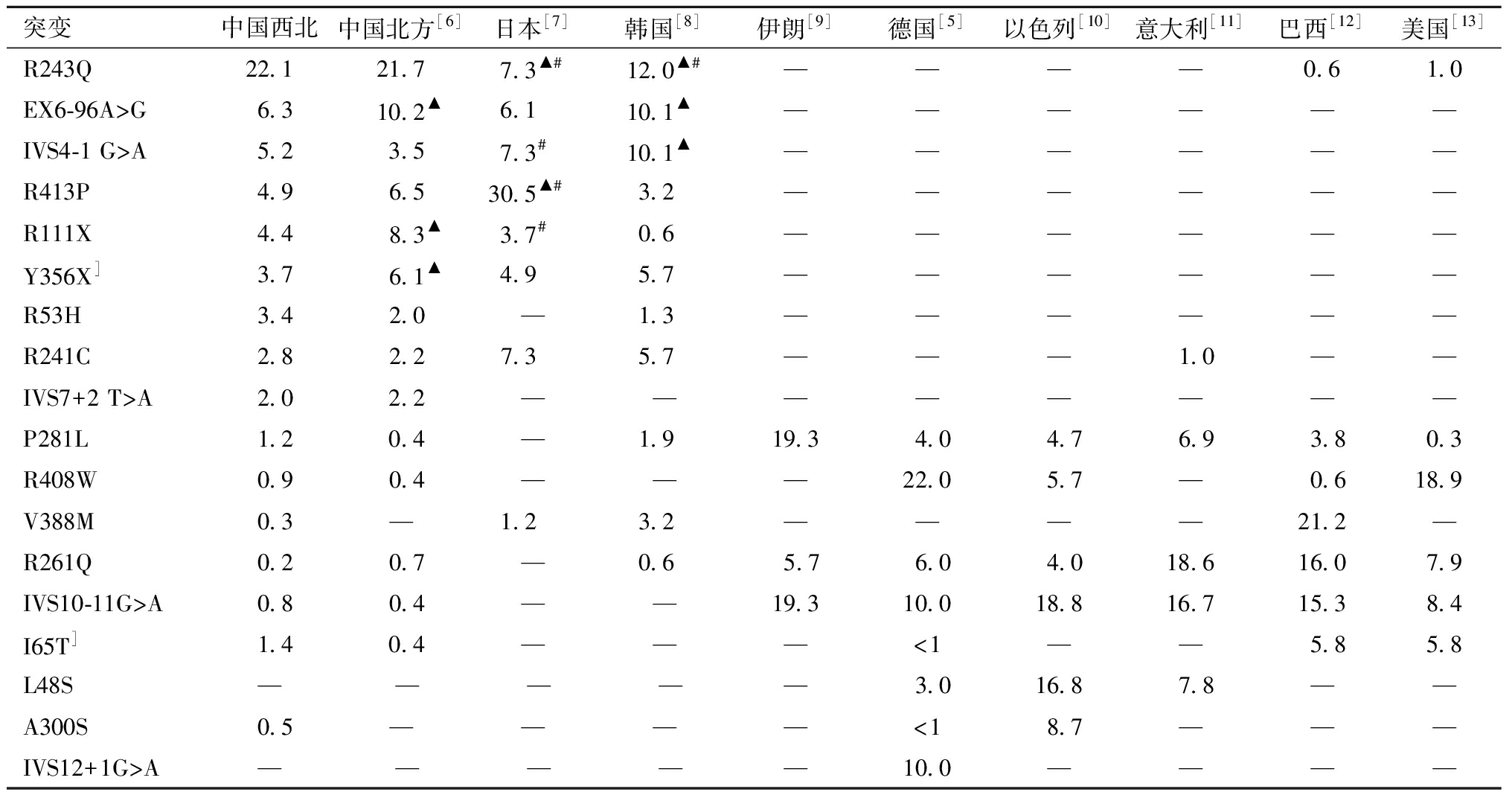
突变中国西北中国北方[6]日本[7]韩国[8]伊朗[9]德国[5]以色列[10]意大利[11]巴西[12]美国[13]R243Q22.121.77.3▲#12.0▲#————0.61.0EX6-96A>G6.310.2▲6.110.1▲——————IVS4-1 G>A5.23.57.3#10.1▲——————R413P4.96.530.5▲#3.2——————R111X4.48.3▲3.7#0.6——————Y356X]3.76.1▲4.95.7——————R53H3.42.0—1.3——————R241C2.82.27.35.7———1.0——IVS7+2 T>A2.02.2————————P281L1.20.4—1.919.34.04.76.93.80.3R408W0.90.4———22.05.7—0.618.9V388M0.3—1.23.2————21.2—R261Q0.20.7—0.65.76.04.018.616.07.9IVS10-11G>A0.80.4——19.310.018.816.715.38.4I65T]1.40.4———<1——5.85.8L48S—————3.016.87.8——A300S0.5————<18.7———IVS12+1G>A—————10.0————
Note:the north of China includes Beijing, Hebei, Inner Mongolia, Jilin, Liaoning and other provinces and cities. - indicates no report. Compared with northwest China, ▲P<0.05. Compared with north China, # P<0.05
四、PAH基因新突变
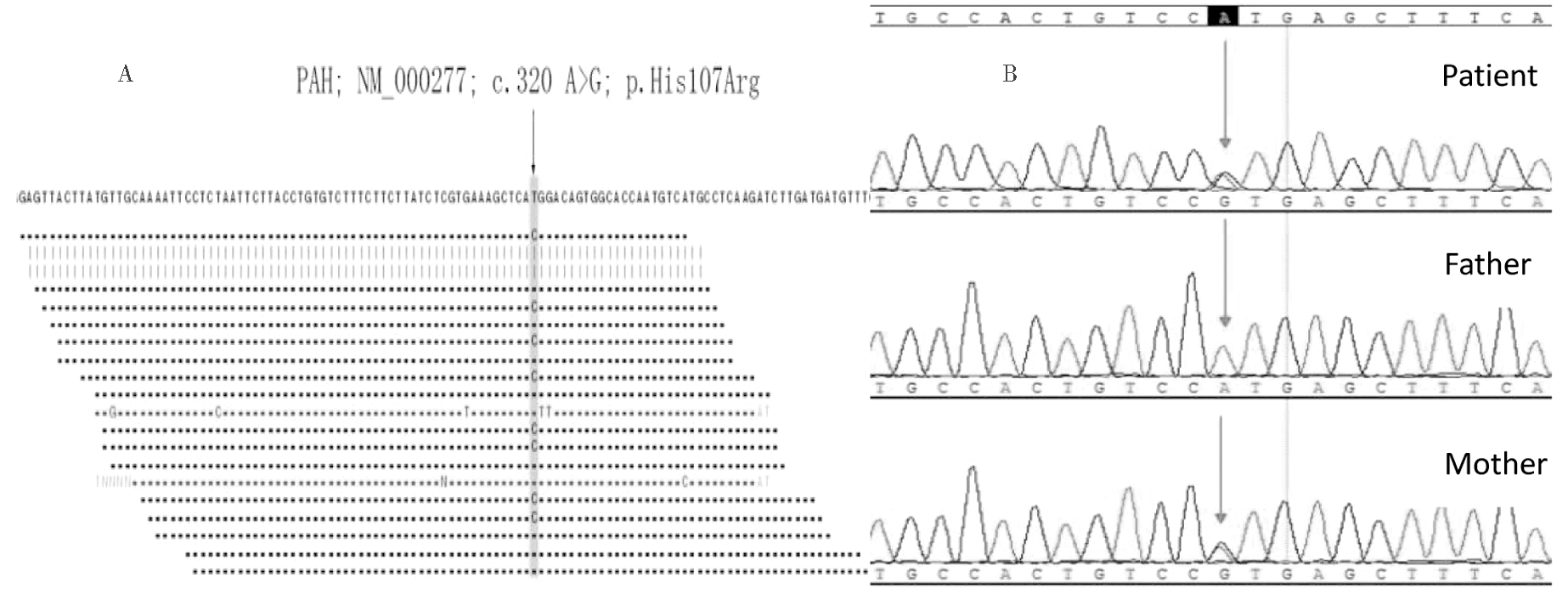
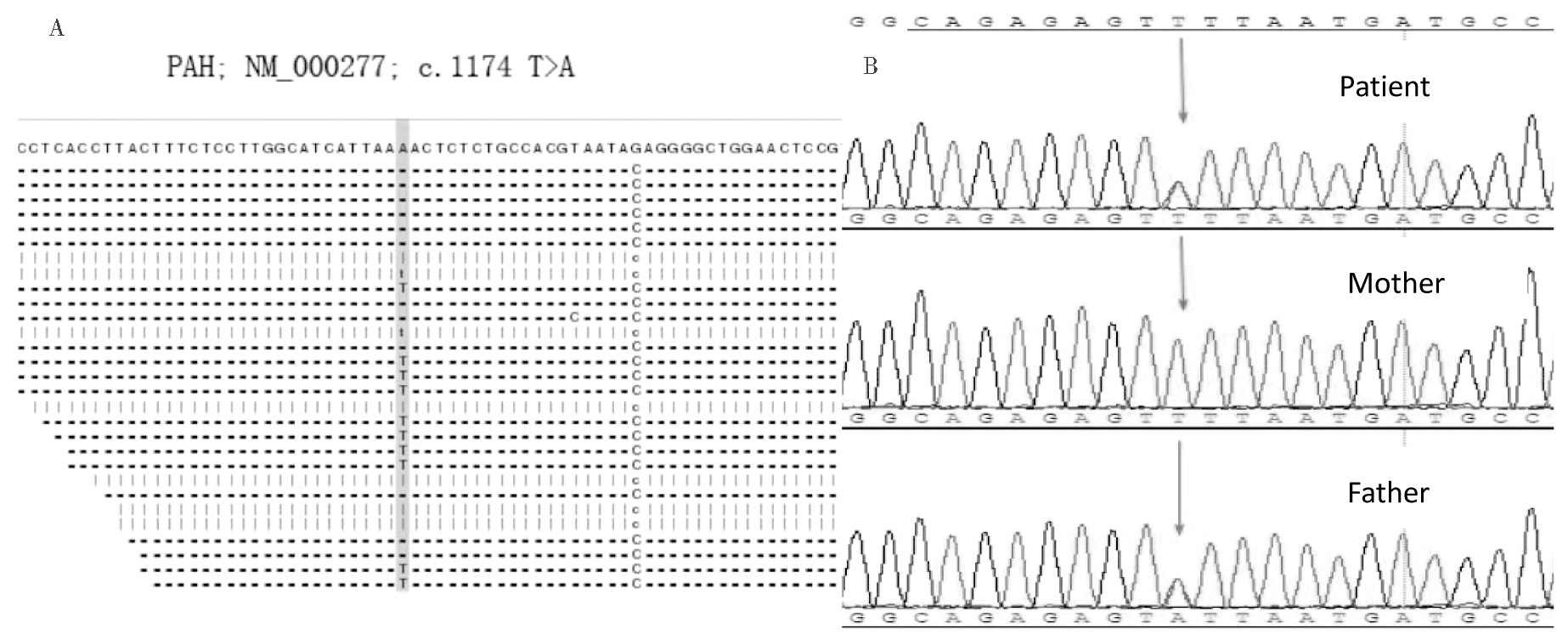
在西北地区检测出的5′-Flanking-626G>C(-626G>C;5′-UTR)、5′-Flanking-480DelACT(-480DelACT;5′-UTR)、M1I(c.3G>T,p.Met1Ile;E1)、S16fsX38(c.47_48delCT,p.Ser16SerfsX10;E1)、N93fsX5(c.279_282delCATC,p.Asn93LysfsX5;E3)、H107R(c.320 A>G,p.His107Arg;E3)、G171E(c.512G>A,p.Gly171Glu;E6)、D222G(c.665A>G,p.Asp222Gly;E6)、F392I(c.1174 T>A,p.Phe392Ile;E11)、N223I(c.668A>T,p.Asn223Ile;E6)、P225S(c. 673C>T,p.Pro225Ser;E6)、Q304K(c.910 C>A,p.Gln304Lys;E8)、IVS11+1G>A(c.1199+1G>A:I11)和L430P(c.1289T>C,p.leu430Pro;E12)这14种突变经检索国内外文献及PAH相关数据库,是本研究在国际上首次报道的PAH基因新突变,并已提交国际PAH突变基因数据库(http://www.biopku.org/pah/)登记入库。
H64fsX9(c.190delC,p.His64ThrfsX9;E3)、T81N(c.242C>A,p.Thr81Asn;E3)、V230A(c.689T>C,p.Val230Ala;E6)、P281L(c.842C>T,p.Pro281Leu;E7)、S303fsX38(c.907delT,p.Ser303ProfsX38;E8)、L348V(c.1042C>G,p.Leu348Val;E10)、P281R(c.842C>G,p.Pro281Arg;E7)、R169C(c.505C>T,p.Arg169Cys;E5)和Y414X(c.1242C>A,p.Tyr414Ter;E12)这9种突变是在中国人群中首次报道的新突变。
H107R突变为PAH基因第3外显子上的错义突变,位于酶单体中具有伸缩性的N末端调节区(1~110残基),该突变可影响酶单体蛋白的催化效率,引起蛋白质的异常折叠、聚合,导致酶功能活性降低(图1);F392I突变为PAH基因第11外显子上的错义突变,位于螺旋富集的催化区(111~410残基),该突变可严重影响苯丙氨酸羟化酶的催化活性,影响正常的单体状态和蛋白酶的功能异常或丧失(图2)。
讨 论
研究发现中国西北地区PAH基因突变的热点区域主要集中于第2、3、5、6、7、11、12外显子及内含子4中(86.5%),与报道的中国北方地区(84.6%)大致相同[7],而与欧洲人更多集中在第3、10和12外显子明显不同[6]。本研究共发现105种PAH基因致病突变,总检出率为87.7%,突变形式主要以错义突变、剪切位点突变、无义突变和移码突变为主(占比92.0%),无论是突变种类数还是突变形式均显著区别于日本[8]、韩国[9]、伊朗[10]、德国[6]、意大利[12]、西班牙[15]、巴西[13]、美国[14]等其他亚洲、欧洲和美洲国家。本地区发现的各种散在的基因突变类型(R158Q、R161Q、L348V、E280K、P281L、R408W等)却在欧洲和拉美地区十分常见,表现出中国西北地区复杂的遗传背景及鲜明的地区特征。
中国西北地区PKU患者中居于首位的PAH基因致病突变为R243Q(22.1%),与中国北方[7]、韩国[9]基本一致,但明显区别于日本最常见的R413P、以色列最常见的IVS10-11G>A和巴西最常见的V388M突变[7,12],也区别于德国[6]、美国[14]等欧美国家最常见的R408W突变,其他常见突变如EX6-96A>G(6.3%)、IVS4-1G>A(5.2%)、R413P(4.9%)等突变在中国北方、日本和韩国等东亚国家较为多见,但却在西亚、欧洲和美洲国家极为少见甚至未有报道,这充分表明中国西北地区PAH基因致病突变类型与中国北方其他省区及东亚国家表现出密切的联系,是中国与欧美地区之间一个较为特殊的PAH突变基因分布带。
R243Q是中国西北汉族、回族中最常见的PAH基因突变,显著区别于西北维吾尔族(R413P),其在西北汉族中的的检出率也显著高于西北回族和西北维吾尔族(P<0.05)。EX6-96 A>G突变在西北回族中的检出率显著低于西北汉族和西北维吾尔族(P<0.05),三个民族的PAH基因常见突变的分布频率具有显著性差异(P<0.05),这充分表明西北少数民族中的PAH突变基因既有密切联系,同时也存在这明显差异,各民族均形成了自身独立的遗传规律和特点[16],推测西北地区R243Q、IVS4-1 G>A突变频率的高处存在于西北汉族中,西北地区R241C、Q232X突变频率的高处存在于西北回族中,R413P、F161S、IVS10-11G>A突变的高处存在于西北维吾尔族中,这对于研究本地区不同民族的遗传流行病学和人类学特点提供了参考依据。
苯丙氨酸羟化酶通常以具有催化活性的四聚体形式存在,四聚体是由两个二聚体组成。根据突变影响苯丙氨酸羟化酶蛋白残基的位置,PAH基因突变可分为大约五种形式:(1)酶活性位点突变;(2)调控区和催化区关键结构突变;(3)调控区和催化区交界出突变;(4)末端调节序列突变;(5)二聚体或四聚体交接处突变[17-18]。本研究在国际上首次报道了14种PAH基因致病突变,其中5′-Flanking-626G>C(错义突变)和5′-Flanking -480DelACT(缺失突变)位于5′非翻译区(启动子区),可造成PAH基因翻译及表达失败,进而影响酶活性降低。M1I、S16fsX38(移码突变)、N93fsX5(移码突变)和H107R这4种突变位于酶单体中具有伸缩性的N末端调节区(1~110残基),而G171E、D222G、N223I、P225S、Q304K和F392I这6种突变均为错义突变,位于螺旋富集的催化区(111~410残基),L430P突变位于C末端的四聚体区(428~452残基),IVS11+1G>A属于第11内含子上的剪切位点突变,这12种突变均位于外显子或外显子与内含子的交接区,严重影响PAH基因转录(含mRNA剪切)、翻译,引起蛋白质的异常折叠、聚合和加速降解,严重影响了苯丙氨酸羟化酶的催化活性,甚至导致酶四聚体结构的高度不稳定,影响正常的单体状态和蛋白酶的功能异常或丧失。
本研究发现中国西北五省区PAH突变基因的检出率为87.7%,与周保成等(96.7%)h和陈雅芬等(95.3%)等国内研究[19-20]相比,突变基因检出率较低,分析认为可能与所采用方法有关,除了PCR产物直接测序法、基因芯片捕获和二高通量测序技术,没有联合采用高分辨熔解曲线和多重连接依赖探针扩增技术,多种技术平台的联合使用可显著提高PAH突变基因的检出率;另一方面,本研究推测本地区遗传异质性复杂,导致某些突变可能存在于所采用技术的检测区域之外,如深度内含子、5′非翻译区、3′非翻译区及与基因表达密切相关的顺式作用元件、反式作用因子以及PAH基因启动子或增强子的序列改变等[21]。
本研究基本明确了中国西北地区PAH基因突变的类型、频率及分布特征,发现本地区具有较为复杂的遗传背景,伴随历史、地理及民族融合等诸多因素影响,人口来源多样繁杂,形成其鲜明的地域和民族特征,是中国与欧亚之间相互区别又相互联系的一个较为特殊的PAH基因分布带,是研究基因突变多样性、PAH基因异质性特点以及人类起源、迁徙、融合等的理想资源地。对于阐述PKU发病的分子机理,丰富及完善中国多民族PAH基因突变谱积累了重要资料,为本地区深入开展PKU的基因诊断、产前诊断,实现优生优育和提高出生人口素质也奠定了坚实理论基础。
参考文献
1 顾学范,王志国.中国580万新生儿苯丙酮尿症和先天性甲状腺功能减低症的筛查.中华预防医学杂志,2004,38:99-102.
2 余伍忠,毛新梅,王慧珍,等.我国西北地区新生儿疾病筛查状况分析.中国优生与遗传杂志,2014,22 :78-79,81.
3 Mitchell JJ,Trakadis YL,Scriver CR.Phenylalanine hydroxylase deficiency.Genet Med,2011,13:697-707.
4 Guldberg P,Romano V,Cerattp NP,et al.Mutational spectrum of phenylalanine hydroxylase defiency in Sicily:implications for diagnosis of hyper pheenylalaninemaia in Southern Europe.Hum Mol Genet,1993,2:1703.
5 Bercovich D,Elimelach A,Zlotogora J,et al.Genotype-phenotype correlations analysis of mutations in the phenylalanine hydroxylase (PAH) gene.J Hum Genet,2008,53:407-418.
6 Johamnes Z.Phenylketonuria mutations in Europe.Hum Mutat,2003,21:345-356.
7 宋昉,瞿宇晋,杨艳玲,等.中国北方地区苯丙氨酸羟化酶基因的突变构成.中华医学遗传学杂志,2007,24:241-246.
8 Okano Y,Asada M,Kang Y,et al.Molecular characterization of phenylketonuria in Japanese patients.Hum Genet,1998,103:613-618.
9 Lee DH,Koo SK,Lee KS,et al.The molecular basis of phenylketonuria in Koreans.J Hum Genet,2004,49:617-621.
10 Mortaza B,Omid O,Shiva MM,et al.Mutations of the phenylalanine hydroxylase gene in Iranian Azeri Turkish patients with phenylketonuria.Genet Test Mol Bioma,2010,14:233-235.
11 Bercovich D,Elimelech A,Yardeni T,et al.A mutation analysis of the phenylalanine hydroxylase (PAH) gene in the Israeli population.Ann Hum Genet,2007,72:305-309.
12 Daniele A,Scala I,Cardillo G,et al.Functional and structural characterization of novel mutations and genotype-phenotype correlation in 51 phenylalanine hydroxylase deficient families from southern Italy.Febs J,2009,276:2048-2059.
13 Santos LL,Castro-Magalhaes M,Fonseca CG,et al.PKU in Minas Gerais State,Brazil:mutation analysis.Ann Hum Genet,2008,72:774-779.
14 Steven F,Clinton E,Thomas C,et al.Mutations in the phenylalanien hydroxylase gene identified in 95 patients with phenylketonuria using novel systems of mutation scanning and specific genotyping based upon thermal melt profiles.Mol Genet Metab,2007,91:218-227.
15 Couce ML,Boveda MD,Femandez-Marmiesse A,et al.Molecular epidemiology and BH4-responsiveness in patients with phenylalanine hydroxylase deficiency from Galicia region of Spain.Gene,2013,521:100-104.
16 毛新梅,何江,刘媛,等.35例回族苯丙酮尿症患儿苯丙氨酸羟化酶基因突变分析.中华医学遗传学杂志,2014,31:6-10.
17 张志,何蕴韶.苯丙酮尿症分子遗传学研究进展.遗传,2004,26:729-734.
18 Erlandsen H,Stevens RC.The stuctural basis of phenylketonuria.Mol Genet Metab,1999,68:103-125.
19 周保成,穆原,尹婷,等.Ion Torrent PGMTM测序仪检测苯丙酮尿症患儿苯丙氨酸羟化酶基因突变。临床检验杂志,2014,32:898-902.
20 陈雅芬,蒋新液,陈忠海,等.中华实用儿科临床杂志,2015,30:1554-1556.
21 余伍忠.PAH基因异质性特点及其突变研究的意义.中国优生与遗传杂志,2006,14:9-10.
(收稿日期:2020-09-02)