子宫内膜癌(uterine corpus endometrial carcinoma,UCEC)是女性生殖系统三大恶性肿瘤之一[1]。UCEC发病率居女性生殖系统恶性肿瘤首位,其发病率逐年上升,并呈年轻化态势。以肿瘤免疫系统为治疗靶点,被认为是抗肿瘤药物研发的一个极具潜力的方向。目前针对UCEC免疫治疗的抗PD-1帕姆单抗,经美国食品药品监督管理局批准已上市[2]。本研究旨在通过生物信息学方法系统地评估UCEC的免疫环境和研究其免疫相关基因。
基于UCEC表达谱数据集评估各肿瘤浸润免疫细胞(tumor infiltrating immune cells,TIICs)的相对比例,探究其与临床特征之间的相关性。其次基于大量数据集,采用生物信息学方法证实UCEC免疫微环境的改变,鉴定影响微环境的核心基因并对该基因介导的作用通路进行富集分析。具体实验设计流程图见图1。
图1 实验总体设计流程图
资料与方法
1.资料来源:从TCGA数据库(https://portal.gdc.cancer.gov/cart)获取UCEC患者的基因表达谱数据、基因突变数据和临床数据。下载406例UCEC组织和19例正常子宫内膜组织的RNA-Seq FPKM数据,以及529例UCEC的SNV突变数据。这些数据包含患者的临床信息,包括发病年龄、肿瘤病理组织学分级、生存状态和以天为单位的生存时间。从GenCode平台(http://www.gencodegenes.org/)中下载人类基因组注释GTF文件。通过免疫学数据库和分析网站(ImmPort; https://www.immport.org/shared/genelists)下载了2 498个与免疫相关的基因,该数据库包含基于各种分子功能的17个免疫类别[3-4]。
2.基于CIBERSORT算法的肿瘤免疫细胞浸润分析:使用R软件CIBERSORT包(版本3.6.3)评估UCEC样本中22种TIICs类型的相对表达水平,即:天然B细胞、记忆B细胞、浆细胞、CD8+T细胞、幼稚CD4+T细胞、CD4+静息记忆T细胞、CD4+激活记忆T细胞、滤泡性辅助T细胞、调节性T细胞、γδT细胞、静息自然杀伤细胞、天然杀伤细胞、单核细胞、M0巨噬细胞、M1巨噬细胞、M2巨噬细胞、静息树突状细胞、活化树突状细胞、静息肥大细胞、活化肥大细胞、嗜酸性粒细胞和嗜中性粒细胞。通过CIBERSORT反卷积算法,将UCEC的mRNA矩阵数据与来自CIBERSORT平台的22种TIICs特征矩阵数据进行比较,分别生成高、低免疫评分组的UCEC组织中的22种TIICs比例矩阵。
3.子宫内膜癌免疫微环境分析:通过ESTIMATE算法对TCGA队列的mRNA矩阵数据进行肿瘤微环境评分[5,6],包括基质细胞评分和免疫细胞评分,根据评分中位值水平将其分为高评分组和低评分组,继而进行差异分析,使用R软件limma包(版本3.8)Wilcox.Test方法分析数据,筛选符合标准的基因。
4.肿瘤突变负荷分析:使用Perl软件(版本v5.30.0)计算每个样本的肿瘤突变负荷(tumor mutation burden,TMB)值,并将UCEC的mRNA矩阵数据按TMB中位值水平分为高TMB组和低TMB组,分析TMB值与生存率的关系。使用Wilcox.Test方法对高、低TMB组的基因进行差异分析,筛选满足条件的基因。
5.获取关键基因:首先对从肿瘤微环境评分分组和TMB分组所获得的差异表达基因进行Venn分析(http://bioinformatics.psb.ugent.be/webtools/Venn/)取交集基因。然后,从ImmPort数据库(https://www.immport.org/shared/genelists)[7]检索免疫相关基因与上述交集基因再次行Venn分析,获得与UCEC相关的候选基因。最后,基于TCGA队列UCEC的临床特征对筛选的基因进行单因素逻辑回归分析[8],筛出关键基因。推测该基因最可能作为UCEC进展中免疫相关的目标基因。
6.基于泛癌分析的靶基因验证:在UCSCXena网站下载TCGA数据库中33种主要的肿瘤转录组数据(FPKM)、突变数据(varscan)和临床数据,使用perl软件提取CXCL14表达矩阵用于下述分析。具体肿瘤见附件表S1。
提取TCGA中各肿瘤的临床数据包括无进展间期生存率、生存状态和生存时间,使用R软件survival、survminer、forestplot包(版本3.6.3)将CXCL14表达矩阵与各样本PFI生存数据合并,根据CXCL14表达量中位水平将样本分为高、低两组进行K-M分析获得PFI生存曲线,最后基于Cox风险比例模型描述基因对生存的影响并绘制森林图。
计算33种肿瘤体细胞突变样本(n=10 114)的TMB值,并提取每个样本(n=10 416)的微卫星不稳定(microsatellite instability,MSI)值,通过spearman方法对CXCL14表达量与TMB值和MSI值进行相关性检验,绘制TMB和MSI相关性雷达图。计算33种肿瘤所有样本的免疫微环境评分,并结合CXCL14在样本中的表达量,检验CXCL14表达量与免疫微环境的相关性。
7.CXCL14基因富集分析(GSEA):为研究筛选的关键基因潜在的分子机制,进行基因富集分析,包括KEGG分析和GO分析。设定GSEA软件重复排列数为1000次,P<0.05。
结果
1. UCEC中的肿瘤免疫微环境:TCGA队列中229例肿瘤样本和8例正常样本符合CIBERSORT P<0.05的筛选标准。UCEC组织中滤泡辅助T细胞(P=0.001)、调节T细胞(P<0.001)、M0巨噬细胞(P<0.001)、浆细胞(P=0.052)的比例与正常组织相比差异具有或临界统计学意义,而相较于正常组织,幼稚B细胞(P=0.005)、CD4+静息记忆T细胞(P=0.001)、γδT细胞(P<0.001)、单核细胞(P=0.01)、M2巨噬细胞(P=0.04)、静息肥大细胞(P<0.001)在UCEC组织中低表达(图2a)。结合患者的生存时间、生存状态、病理组织学分级及年龄分析,发现调节T细胞(P=0.031)高丰度与患者生存率正相关,调节T细胞(P<0.001)、CD4+静息记忆T细胞(P<0.001)高丰度与UCEC病理组织学分级负相关,静息肥大细胞(P=0.015)丰度在年龄≤60岁患者组与年龄>60岁组中差异具有统计学意义,浆细胞(P=0.032)丰度与年龄的关系与静息肥大细胞相反(图2b、c、d、e)。此外,免疫微环境基因经差异分析获得549个上调基因和1个下调基因。
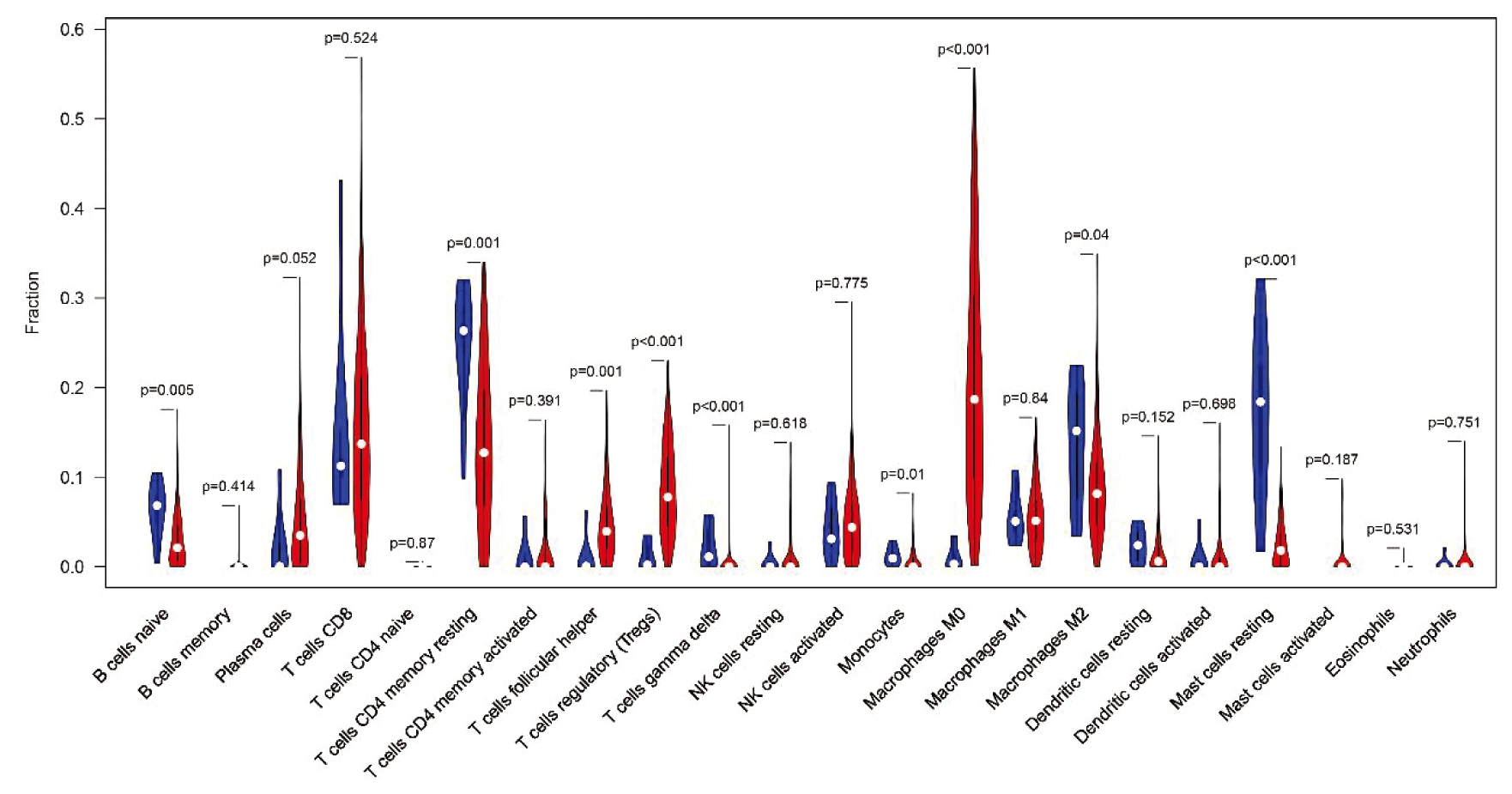
图2a TIICs在子宫内膜正常组织和癌组织中的分布和丰度差异
注:红色为癌组织,蓝色为正常组织。
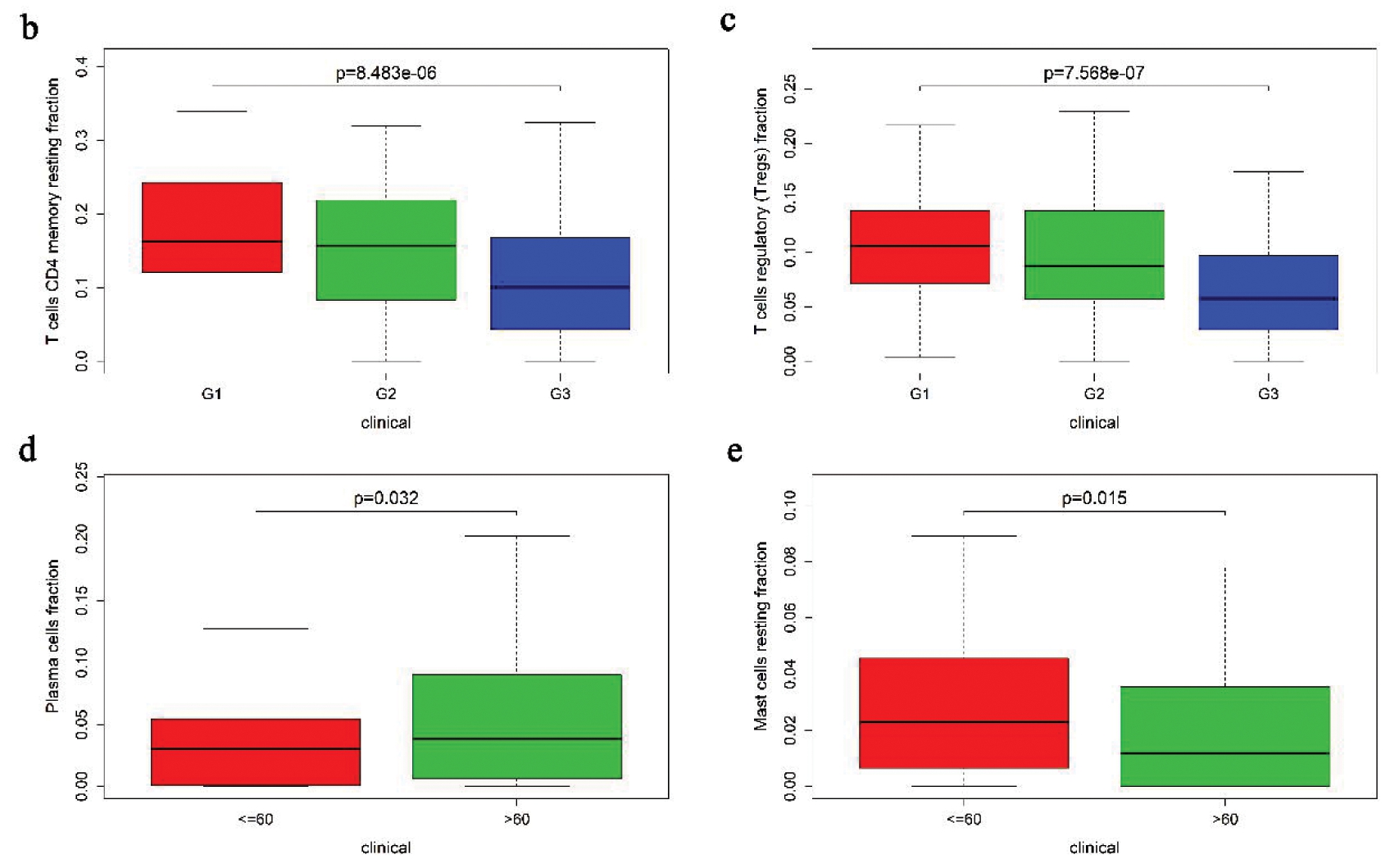
图2b CD4+静息记忆T细胞丰度与患者病理分级的相关性;图2c 调节T细胞丰度与患者病理分级的相关性;图2d 浆细胞丰度与患者年龄的相关性;图2e 静息肥大细胞丰度与患者年龄的相关性
注:UCEC按照病理组织学分级分为高分化(G1) 、中分化(G2) 、低分化(G3);“<=60”为小于或等于60岁的患者,“>60” 为大于60岁的患者。
2. UCEC肿瘤突变负荷:将UCEC的mRNA矩阵数据TMB值按中位值水平分为高TMB组(n=226)和低TMB组(n=165),结合生存时间及病理组织学分级分析发现,高TMB值组生存率与低TMB值组相比差异具有统计学意义(P=0.023),且高TMB值与UCEC患者病理组织学分级呈正相关(P<0.001,图3)。此外,对UCEC表达谱基因高、低TMB值组进行基因差异分析获得326个差异基因。
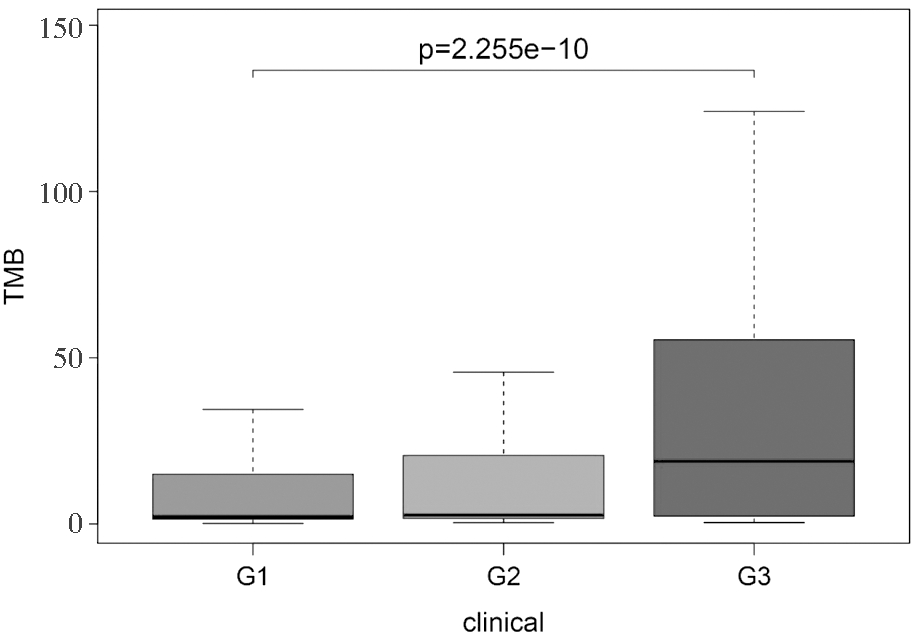
图3 TMB值与患者病理分级的相关性结果示意图
注:UCEC按照病理组织学分级分为高分化(G1) 、中分化(G2) 、低分化(G3)
3. UCEC免疫微环境中与预后相关的关键基因:对肿瘤免疫微环境分组获得的392个差异基因和TMB值分组获得的326个差异基因以及在ImmPort数据库检索到的2498个免疫相关基因进行Venn分析获得9个重叠基因(图4a)。最后,基于上述9个基因表达量的差异筛选出与免疫微环境关系最密切的基因CXCL14,CXCL14的表达量在肿瘤样本中明显高于正常样本(P<0.001)(图4b)。结合406例UCEC样本的临床特征分析发现,CXCL14在年龄≤60岁组中表达量显著高于年龄>60岁者,其低表达与病理组织学分级正相关(P<0.05,图4c、d)。
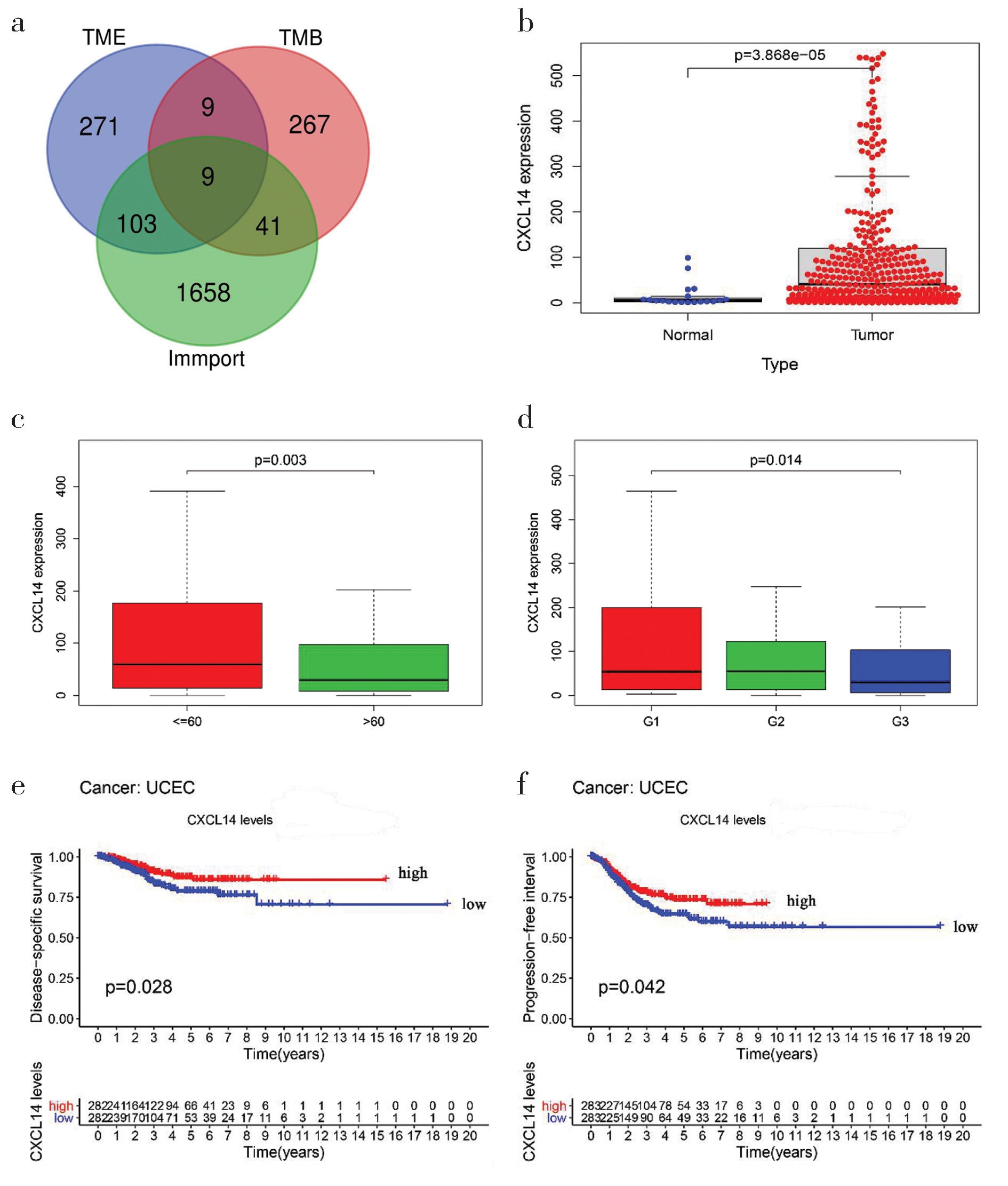
图4a 基于生物信息学网站获得免疫微环境、TMB和免疫相关基因的Venn图;图4b CXCL14在正常样本和肿瘤样本中表达量示意图;图4c CXCL14在406例肿瘤样本中的表达量与年龄的相关性;图4d CXCL14在406例肿瘤样本中的表达量与病理分级的相关性;图4e CXCL14的表达与患者疾病特异生存率的关系;图4f CXCL14的表达与患者无进展间期生存率的关系。
(注:UCEC按照病理组织学分级分为高分化(G1) 、中分化(G2) 、低分化(G3)。“<=60”为小于或等于60岁的患者;“>60” 为大于60岁的患者。)
在UCEC中CXCL14的表达量与疾病特异生存率(disease specific survival,DSS)和无进展间期生存率(progression free interval,PFI)具有相关性。基于R语言limma包,采用Kaplan-Meier法获得了DSS、PFI生存曲线,发现CXCL14低表达患者的疾病特异生存率明显低于高表达患者(P=0.028,图4e),并且CXCL14高表达患者无进展间期生存率显著高于低表达者(P=0.042,图4f)。
4. CXCL14在33种肿瘤中的表达:对33种肿瘤共11 057例样本(T=10 327,N=730)的CXCL14表达矩阵进行差异分析,发现CXCL14在子宫内膜癌、宫颈鳞癌等16种肿瘤中的正常组织和癌组织之间的差异表达有意义(P=0.001 "***" ,P=0.01"**" ,P=0.05"*",P=1" ",图5a)。采用COX分析方法探究CXCL14在肿瘤中的表达量与总生存时间的相关性,结果表明CXCL14在UCEC等12种肿瘤中的表达与患者总生存时间相关,且CXCL14为低风险基因(图5b)。
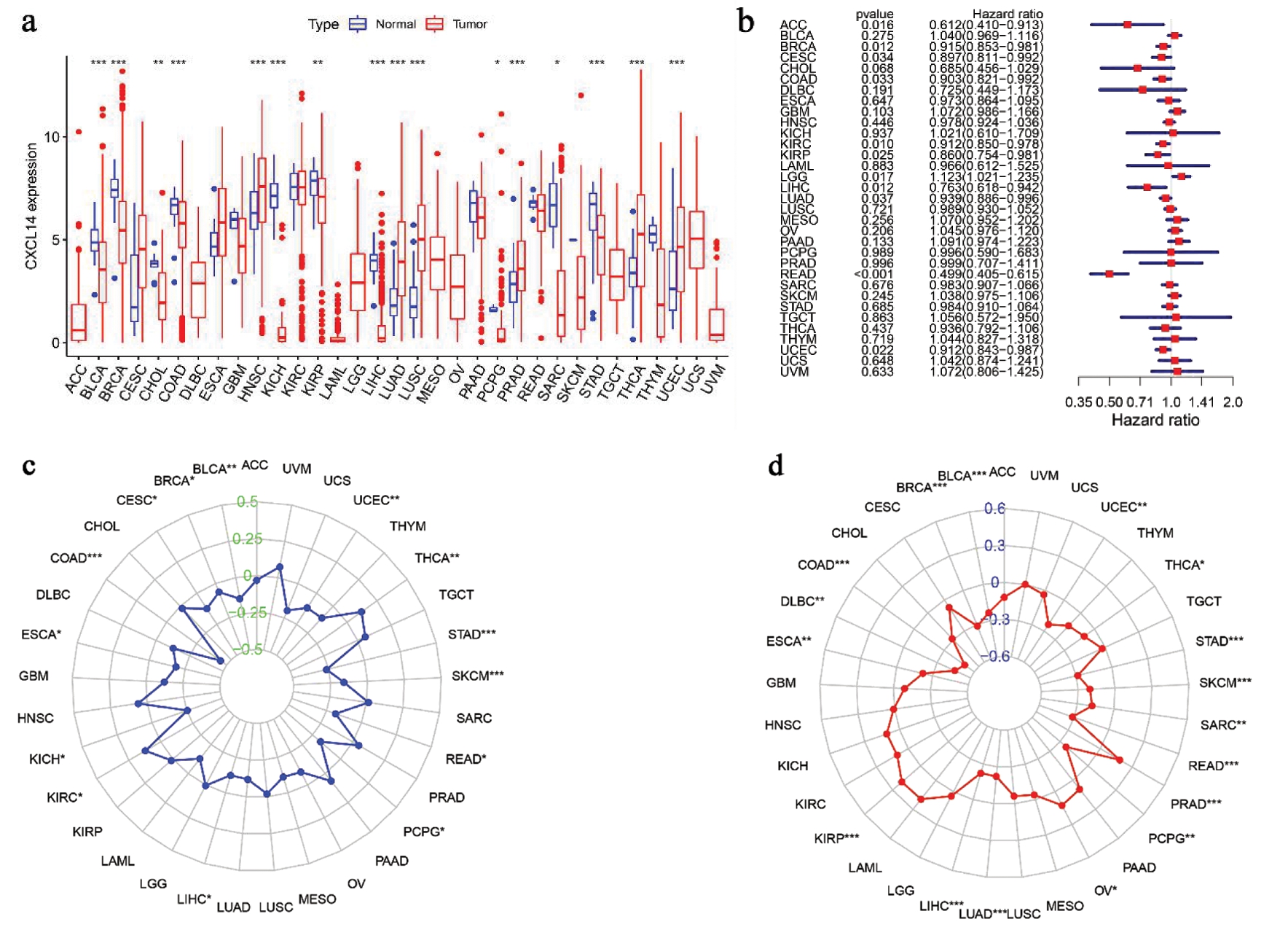
图5a CXCL14在33种肿瘤中的正常组织和癌组织之间的表达差异;图5b CXCL14在肿瘤中的表达量与总生存时间的相关性;图5c 33种肿瘤中CXCL14的表达量与TMB相关性示意图;图5d 33种肿瘤中CXCL14的表达量与MSI相关性示意图
包括UCEC在内的17种肿瘤的CXCL14表达量与TMB值具有相关性;包括UCEC在内的12种肿瘤的MSI值与CXCL14的表达负相关(P=0.001 "***" ,P=0.01"**" ,P=0.05"*",P=1" ",图5c、d)。
5.CXCL14富集生物通路:CXCL14基因集低表达富集203例,高表达富集203例。由KEGG分析可知,CXCL14低表达富集会抑制B细胞受体信号通路、错配修复、DNA复制、泛素介导的蛋白水解作用等通路(图6)。由富集分析可知,CXCL14在UCEC中是一个保护性基因,其下调会抑制体内正常的免疫反应,从而使癌细胞逃避免疫过程。
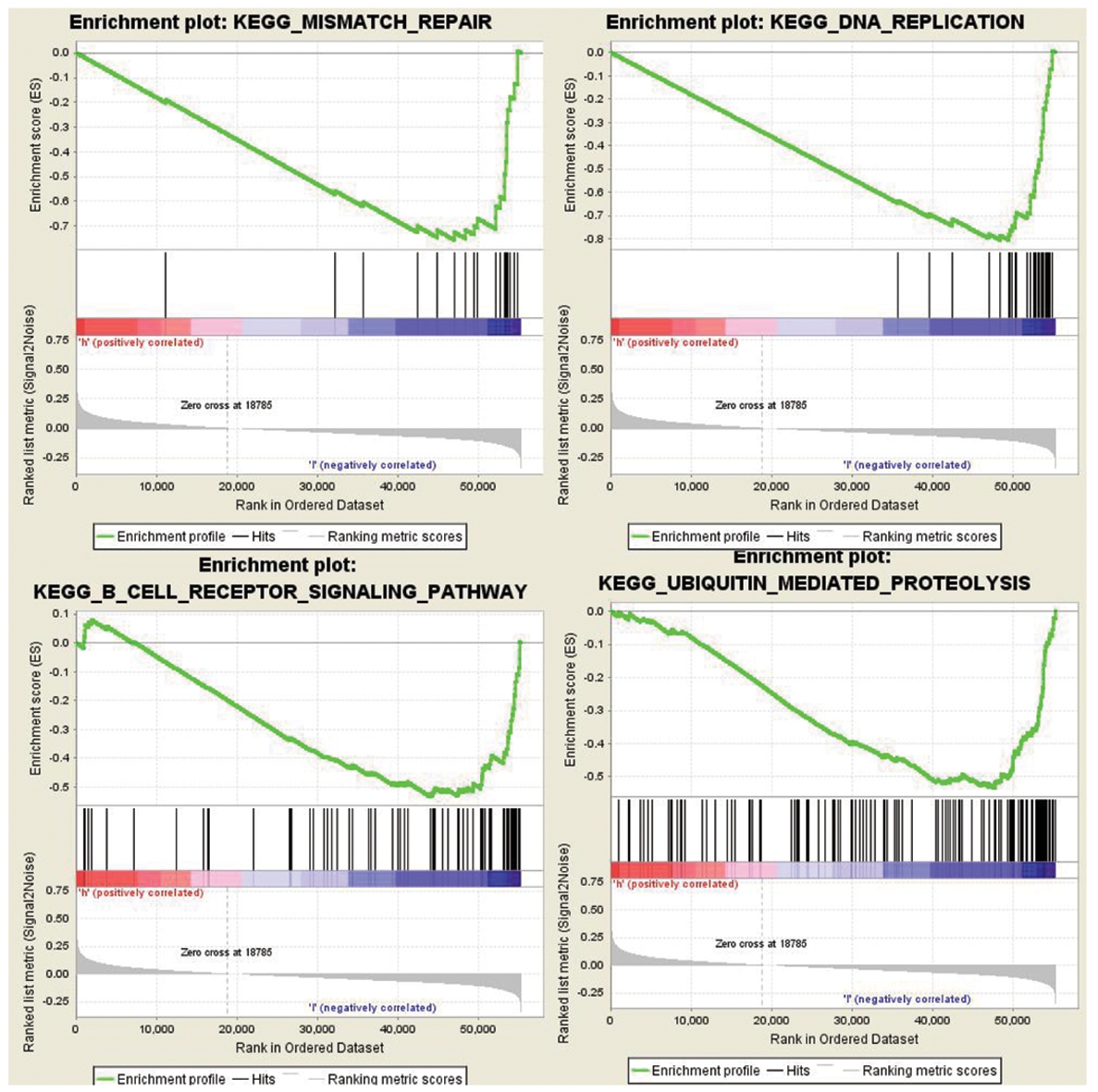
图6 CXCL14在UCEC有关通路中的富集示意图
讨论
研究表明UCEC间质中有大量免疫细胞浸润[9-10],免疫细胞浸润被认为在多种恶性肿瘤的发生发展起着重要作用[5,11],目前免疫治疗在抗肿瘤领域取得了巨大进展。本研究发现,UCEC中浸润的免疫细胞比例高于正常组织,部分免疫细胞浸润丰度与患者预后相关,针对这些免疫细胞的靶向治疗,有望改善患者预后。
近年来免疫系统疗法如免疫检查点抑制剂等在抗肿瘤领域效果显著。研究表明肿瘤基因高度突变可诱导产生大量新抗原,从而激活免疫细胞,导致机体产生抑制肿瘤的免疫反应[12]。MSI与肿瘤免疫治疗的疗效关系密切[13],多项研究已证实TMB是预估肿瘤免疫治疗疗效的新兴生物标志物[14]。本研究发现UCEC高TMB组生存率明显高于低TMB组,而TMB值越高病理分级越高,我们推测高级别UCEC患者有望通过免疫治疗获益。
CXCL14是一种高度保守的、稳态的趋化因子,负责免疫细胞的募集和成熟,可作为抗微生物和抗肿瘤因子发挥作用[15]。CXCL14在宫颈癌、结直肠癌、子宫内膜癌和头颈癌等癌症中经常出现失调,其破坏被证明限制了关键的抗肿瘤免疫调节[16]。有研究证实,CXCL14通过抑制肿瘤血管的生成起到抑制肿瘤发展的作用[17]。本研究发现CXCL14在UCEC中的表达量高于正常组织,且CXCL14的表达与病理分级、患者生存率及年龄相关,推测CXCL14表达失调可能与UCEC患者不良预后有关。这提示CXCL14不仅是判断UCEC预后的重要指标,而且是一个新的潜在靶标。在不同类型肿瘤中CXCL14的表达不一致,如在肾癌、肺癌、头颈癌、宫颈癌等组织中几乎不表达或低表达,而在部分前列腺癌和乳腺癌中表达水平却很高[18],这与上述泛癌分析结果一致,CXCL14在肿瘤中的表达可能受多种因素影响。
本研究通过探究UCEC相关免疫细胞浸润状态,发现其在UCEC进展中与免疫过程具有强烈的相关性,为研究UCEC免疫机制提供了理论支持。结合TCGA和ImmPort数据库筛选出参与UCEC进展的相关基因,CXCL14是其中之一。CXCL14在肿瘤中低表达会促进原发性免疫缺陷、癌症发展等,其可作为UCEC有效的免疫相关预后标志物。本研究实验数据来源于TCGA数据库,有待进一步实验来证实。
1 McAlpine J,Leon-Castillo A,Bosse T.The rise of a novel classification system for endometrial carcinoma,integration of molecular subclasses.J Pathol,2018,244:538-549.
2 Brooks RA,Fleming GF,Lastra RR,et al.Current recommendations and recent progress in endometrial cancer.CA Cancer J Clin,2019,69:258-279.
3 Chaussabel D,Baldwin N.Democratizing systems immunology with modular transcriptional repertoire analyses.Nat Rev Immunol,2014,14:271-280.
4 Li S,Rouphael N,Duraisingham S,et al.Molecular signatures of antibody responses derived from a systems biology study of five human vaccines.Nat Immunol,2014,15:195-204.
5 Yoshihara K,Shahmoradgoli M,Martínez E,et al.Inferring tumour purity and stromal and immune cell admixture from expression data.Nat Commun,2013,4:2612.
6 Shah N,Wang P,Wongvipat J,et al.Regulation of the glucocorticoid receptor via a BET-dependent enhancer drives antiandrogen resistance in prostate cancer.Elife,2017,6.
7 Bhattacharya S,Andorf S,Gomes L,et al.ImmPort:disseminating data to the public for the future of immunology.Immunol Res,2014,58:234-239.
8 Liu GM,Zeng HD,Zhang CY,et al.Identification of a six-gene signature predicting overall survival for hepatocellular carcinoma.Cancer Cell Int ,2019,19:138.
9 Liu J,Nie S,Wu Z,et al.Exploration of a novel prognostic risk signatures and immune checkpoint molecules in endometrial carcinoma microenvironment.Genomics,2020,112:3117-3134.
10 Dossus L,Lukanova A,Rinaldi S,et al.Hormonal,metabolic,and inflammatory profiles and endometrial cancer risk within the EPIC cohort-a factor analysis.Am J Epidemiol,2013,177:787-799.
11 Hanahan D,Coussens LM.Accessories to the crime:functions of cells recruited to the tumor microenvironment.Cancer Cell,2012,21:309-322.
12 Büttner R,Longshore JW,López-Ríos F,et al.Implementing TMB measurement in clinical practice:considerations on assay requirements.ESMO Open,2019,4:e000442.
13 Baretti M,Le DT.DNA mismatch repair in cancer.Pharmacol Ther,2018,189:45-62.
14 Samstein RM,Lee CH,Shoushtari AN,et al.Tumor mutational load predicts survival after immunotherapy across multiple cancer types.Nat Genet,2019,51:202-206.
15 Yang XY,Ozawa S,Kato Y,et al.C-X-C motif chemokine ligand 14 is a unique multifunctional regulator of tumor progression.Int J Mol Sci,2019,20.
16 den Boon JA,Pyeon D,Wang SS,et al.Molecular transitions from papillomavirus infection to cervical precancer and cancer:Role of stromal estrogen receptor signaling.Proc Natl Acad Sci U S A,2015,112:E3255-3264.
17 Penk A,Baumann L,Huster D,et al.NMR and molecular modeling reveal specificity of the interactions between CXCL14 and glycosaminoglycans.Glycobiology,2019,29:715-725.
18 Fazi B,Proserpio C,Galardi S,et al.The Expression of the Chemokine CXCL14 correlates with several aggressive aspects of glioblastoma and promotes key properties of glioblastoma cells.Int J Mol Sci,2019,20.