葡萄胎(hydatidiform mole,HM)属于滋养细胞疾病(gestational trophoblastic disease,GTD)中的一种,以绒毛变性水肿、滋养细胞增生为主要特征,主要临床表现为异常升高的β-hCG血值,B超表现为“雪花状”影像[1],根据组织病理学特征大体可以将HM分为完全性葡萄胎(complete hydatidiform mole,CHM)和部分性葡萄胎(partial hydatidiform mole,PHM)两大类。在分子遗传学方面,大部分CHM是由于母源性遗传物质缺如导致的孤雄二倍体,PHM则为多精入卵造成的双亲多倍体,其中CHM中的亚类——双亲型HM(biparental HM,BiHM)是双亲来源的二倍体,考虑是由于HM致病基因如NLRP7和KHDC3L突变引起,有研究发现NLRP7/KHDC3L突变和印迹基因的表达异常有关[2]。西方国家中HM的发病率为1/1 500,亚洲HM的发病率为1/500[3],中国HM患者的发病率为1.00‰~8.83‰[4]。复发性葡萄胎(recurrent hydatidiform mole,RHM)是指同一个患者有两次及以上的葡萄胎病史[5]。HM的再发风险较高,发生第二次HM的风险是1%~2%,而发生第三次HM的风险为15%~20%[5]。同时,HM也有发展成妊娠滋养细胞性肿瘤的可能[6],CHM有15%~20%的恶化可能,PHM的恶化可能低于5%[[5]。HM的再发可能和癌变风险给患者和家属带来了沉重的精神和经济负担。
为了降低HM对患者造成的伤害,保护患者的生育力,越来越多的研究尝试通过胚胎植入前遗传学检测(preimplantation genetic testing,PGT)规避HM的发生风险。分别有研究通过FISH[7-8],STR(short tandem repeat)基因分型[9]、CNV(copy number variant)和单核苷酸多态性微阵列芯片技术(single nucleotide polymorphism array,SNP array)[10]对植入前胚胎进行检测,选择双亲来源的二倍体胚胎进行移植,来排除母源性遗传物质缺如或多精入卵造成的HM类型。然而,目前尚没有基于二代测序(next-generation sequencing,NGS)技术的PGT治疗应用于RHM患者。本研究旨在探究NGS技术在非BiHM型且不携带NLRP7/KHDC3L致病变异的RHM患者胚胎中遗传学检测的应用可行性。
对象与方法
一、对象
本研究纳入了2019—2021年北京大学第三医院生殖中心行辅助生殖技术支持的2例RHM患者夫妇发育停滞的3枚胚胎,其中患者1(RHM1)有1个胚胎,患者2(RHM2)有2个胚胎,收集患者助孕治疗周期中发育停滞至Day5/Day6的胚胎并进行检测。
纳入标准:在行助孕治疗之前RHM患者至少有1次HM清宫组织经STR基因分型明确为非BiHM类型;患者及其配偶经基因检测均不携带NLRP7和KHDC3L可疑致病突变;患者及其配偶为非近亲结婚。
所有入组患者及其家属均充分知情,并签署了书面知情同意书。本研究通过了北京大学第三医院生殖医学伦理委员会的伦理审查。
二、方法
1.提取样本DNA:收集RHM患者及其配偶的血样并用Qiagen试剂盒(QIAamp DNA Mini Kit, 51104)提取DNA;收集患者助孕治疗周期中发育停滞至Day5/Day6的胚胎,将细胞团用口吸管吹散为单个细胞,将2~4个细胞分别转移到单细胞全基因组扩增试剂盒(MALBAC©单细胞扩增剂盒,亿康)的细胞裂解液中,准备进行单细胞DNA扩增。
2.二代测序分析方法:将患者及其家属的DNA,废弃胚胎的单细胞DNA经MALBAC扩增后用NEB试剂盒(NEBNext© ultra DNA library Prep Kit for Illumina©, NEB#E7645S)进行建库,并在Illumina Hiseq Xten平台进行测序,测序的平均深度约为2×。测序数据经过trim_galore软件(0.6.6版本)去除接头和低质量数据,长度大于36 bp的序列保留,然后用BWA软件(0.6.1版)比对到人类参考基因组(hg38版本)。比对后的数据用readCounter计算样本中每个1M窗口的reads数,用HMMcopy(0.1.1版)软件矫正reads数的GC含量和比对率,然后使用HMM方法确定各个样本全基因组的CNV。同时,将比对后的数据用GATK(3.4版)软件计算患者及其配偶和废弃胚胎全基因组的杂合SNP的数量,将各个样本中的杂合SNP数量进行测序深度归一化处理后进行比较。
3.STR基因分型分析方法:将患者及其配偶的DNA,单细胞全基因组经MALBAC扩增后的DNA约0.25~1 ng用PowerPlex© 16HS系统对STR位点进行PCR扩增,并根据患者及其家属各STR位点重复序列的次数不同,来判定胚胎染色体的亲本来源。
结 果
一、应用二代测序技术进行胚胎亲本来源和倍型分析
本研究收集了2例患者发育停滞至Day5/Day6的废弃胚胎,共3枚,患者1(RHM1)有1个胚胎,患者2(RHM2)有2个胚胎。经NGS测序后进行了杂合SNP数量和CNV分析,分别以男女方常染色体上的杂合SNP数量为参照,将二者常染色体上的杂合SNP数量与胚胎常染色体上的杂合SNP数量对比,由于性别会造成X染色体上杂合SNP数量有较大差异,故本研究仅考虑常染色体上的杂合SNP数量。由于所有样本都为MALBAC扩增后的DNA产物,正常的胚胎比值有一定的浮动范围,如果胚胎染色体为单体或单亲二体,杂合SNP数量应该极少,比值会异常增加,需结合CNV结果综合判断。
如图1所示,发现RHM1患者男女方常染色体上的杂合SNP数量与胚胎F1常染色体上的杂合SNP数量比值均在1左右,即患者男女方和胚胎常染色体上的杂合SNP数量相当,考虑到患者男女方均为双亲来源,则可认为F1也为双亲来源,而在CNV图谱(图2)中可以看到,F1在8号染色体上为三体。在RHM2患者中,发现胚胎F1中的2号、4号、8号、18号、20号染色体上,男女方的杂合SNP数量与胚胎F1中的杂合SNP数量的比值异常升高(图1),即胚胎F1中的杂合SNP数量异常降低,考虑为单亲来源,而在CNV图谱(图2)中可以看到,2号、8号、18号、20号染色体为单体,与杂合SNP数量的结论一致,而4号染色体则考虑为单亲来源二体。此外,CNV结果提示胚胎的X染色体为三体。在RHM2患者的胚胎F2中,杂合SNP数量未显示明显异常,而CNV图谱提示胚胎可能存在嵌合现象。

图1 患者及其配偶和胚胎杂合SNP数量的比值
Figure 1 Heterozygous SNP numbers for patients RHM1 (A) and RHM2 (B)

图2 患者RHM1和RHM2胚胎的CNV图谱
Figure 2 CNV in embryos for patients RHM1 (A) and RHM2 (B)
二、STR基因分型方法验证NGS技术应用的可靠性
本研究采用STR基因分型的方法进行验证,发现在RHM1患者中(图3A),F1胚胎在D8S1179位点处有三个峰,且发现两个峰的信号与女方相同,一个峰信号与男方相同,考虑到该位点在人8号染色体上,结合NGS检测结果,即可认为F1胚胎的8号染色体为三体,其中2条染色体来自于患者,1条染色体来自于患者配偶。
本研究在患者RHM2中验证了F1胚胎位于2号染色体上的TPOX位点,4号染色体上的FGA位点和8号染色体上的D8S1179位点和性染色体上的Amelogenin位点(图3B)。发现F1胚胎在TPOX位点处为单峰,且TPOX位点处的单峰在患者RHM2及其配偶中均有,故无法判断其亲本来源。FGA位点处有1个峰信号与患者配偶相同,还有一个杂峰,可认为F1胚胎的4号染色体为父亲来源。D8S1179位点处有一个来源于患者配偶的峰信号和一个杂峰信号,即可认为8号染色体也为父亲来源。Amelogenin位点仅可判断该胚胎的性染色体中不含有Y染色体。而在患者RHM2的F2胚胎中,验证了3个位点发现均为双亲来源,如图3C所示,该结果也与杂合SNP数量的分析结果相符,而3个STR位点D5S818、D13S317、D16S539的结果在判断该胚胎染色体是否存在嵌合现象方面存在一定的局限性。
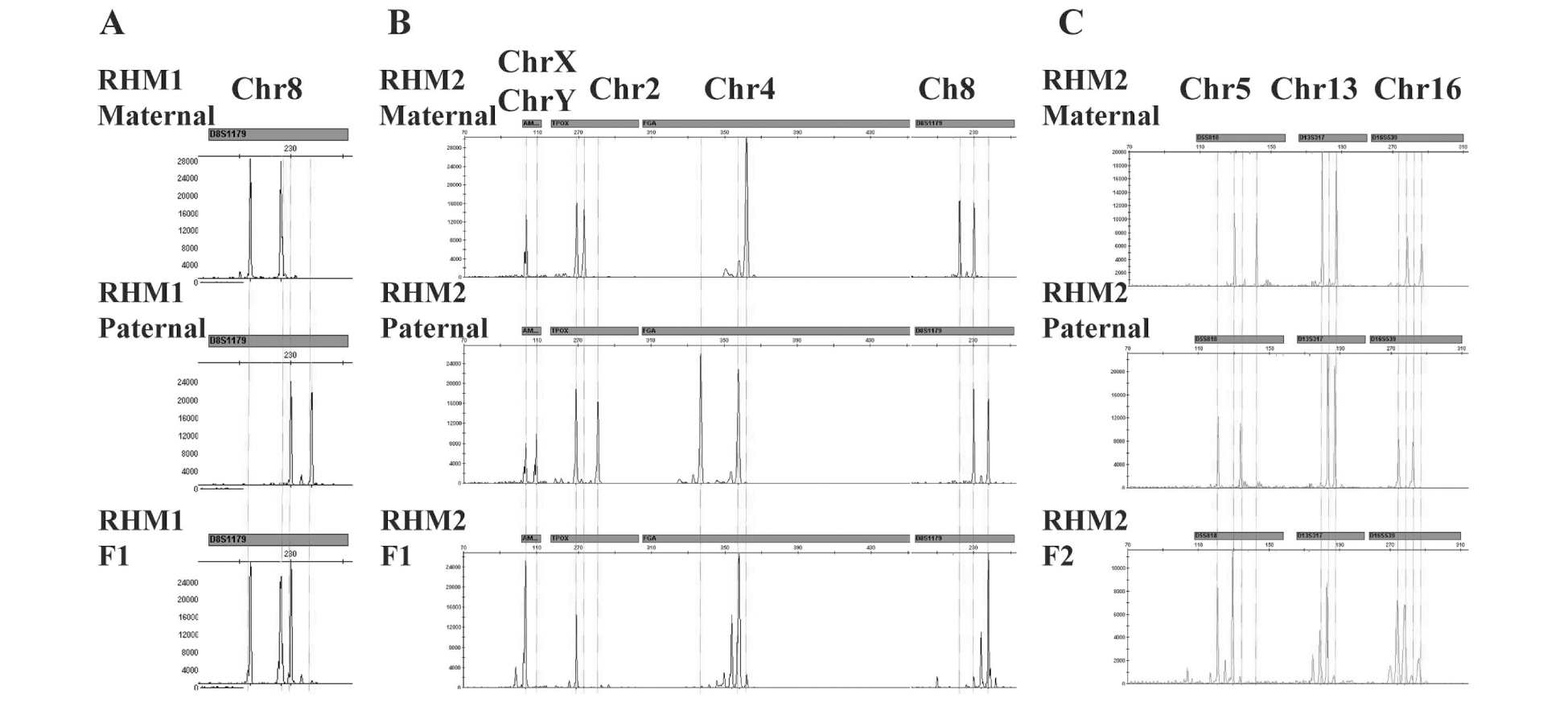
图3 患者RHM1胚胎F1和患者RHM2胚胎F1,F2的STR基因分型验证
Figure 3 The verification of STR genotyping for embryo F1 from patient RHM1 and for embryos F1 and F2 from patient RHM2
讨 论
HM较高的再发风险和癌变可能使得辅助生殖治疗对于HM患者十分必要,然而,一方面由于患者的临床样本较难获得,另一方面由于技术发展受限,既往研究报道中关于PGT技术在RHM患者中的应用较少,本研究利用RHM患者体外发育停滞至Day5/Day6的废弃胚胎进行检测,尽量规避了研究对RHM患者的影响,旨在探究基于NGS技术的染色体亲本来源和倍型分析在RHM患者PGT进程中应用的可行性。
既往应用于RHM患者的PGT技术包括基因分型和NGS技术。1997年有研究第一次将FISH技术应用于复发型妊娠滋养细胞疾病的患者的PGT流程中,该研究在PGT进程中用FISH技术对18、X、Y染色体进行分析,筛选出二倍体的男性胚胎进行移植,然而该研究中的患者移植2枚胚胎后仍未成功受孕[7]。2009年有研究报道了通过对植入前胚胎进行基因分型的方法[9],该研究中的患者有2次雄源性CHM病史,该患者促排后获卵14枚,经卵细胞浆内单精子显微注射技术(intracytoplasmic sperm injection,ICSI)后获得11枚双原核受精卵,在胚胎体外发育至第三天进行活检,活检得到的单个卵裂球通过多重置换扩增(multiple displacement amplification,MDA)技术进行全基因组扩增,该研究中旨在检测分布在13、18、21号染色体上的16个STR位点,实际检测到了10个STR位点用于检测亲本来源;该研究中患者移植1枚胚胎后成功分娩同卵双胎。2017年报道了在RHM患者中进行NGS和SNP array技术应用[10],参与该研究的患者有5次HM病史,然而其HM分型未知;该研究中检测到患者携带有NLRP7 c.2810+2T>G纯合突变,其丈夫未携带NLRP7相关的致病或可疑致病突变位点;该患者促排后获卵15枚,经ICSI后获得9枚双原核受精卵和1枚0PN,10枚胚胎均发育停滞到Day3-Day6,利用SNP array和CNV分析的方法对胚胎进行检测,发现10枚胚胎中仅有2枚胚胎核型正常,其余有7枚胚胎有不同的染色单体异常,1枚胚胎有复杂的染色体异常,但是未发现有单亲来源的胚胎。
既往研究通过不同方法对PGT在RHM患者中的应用进行了尝试,且不同技术方法各有不同。本研究以NGS技术为基础分析了植入前胚胎的杂合SNP数量和CNV,并将其应用于PGT进程中。SNP位点分布于人类基因组中的编码区和非编码区,且以等位基因的形式分布,常用于亲缘关系鉴定,这也是本研究将杂合SNP数量用于判定亲本来源的理论基础[11]。CNV是人类基因组中较常见的染色体结构变异,包括DNA的重复、插入和缺失等,片段长度多大于50 bp,常用于研究染色体的非整倍体型异常[12]。在本研究中,低深度的NGS测序技术可以覆盖全基因组并进行杂合SNP数量和CNV的检测,以此进行亲本来源和染色体倍型分析。由于常染色体上的杂合SNP数量在个体间存在差异,本研究将男女方的杂合SNP数量作为参照,与胚胎的杂合SNP数量对比,由于男女方均为正常的双亲来源二倍体,则双亲来源的二倍体胚胎中杂合SNP数量应该与男女方相近,男女方杂合SNP数量与胚胎杂合SNP数量的比值异常增高,则考虑胚胎为单亲来源,与此同时,也可根据CNV图谱判定胚胎的染色体倍型异常。本研究正是利用杂合SNP数量区分单亲来源或双亲来源,并用CNV可以筛查染色体的异常倍型。遗憾的是,本研究中没有发现单亲来源的二倍体胚胎,但是本研究利用此方法在患者RHM1和患者RHM2的胚胎中筛查出了染色单体,也发现患者RHM2的胚胎F1中的4号染色体为单亲来源的二体,该阳性结果也进一步论证了本研究方法的可靠性。
同时,本研究采用STR基因分型验证了胚胎染色体的亲本来源,发现经STR基因分型验证的结果与NGS检测结果基本吻合,这也进一步证实了基于NGS技术为基础的亲本来源和倍型分型的可信度。STR位点是长度约2~6 bp的短串联重复序列,其重复单元的数量由于高度多态性可以用于区分识别个体[13],STR基因分型正是利用STR位点重复次数的不同来区分亲本来源,但是将STR基因分型直接用于PGT流程中仍有一定的局限性。一方面由于STR位点个数有限,不能有效覆盖全部染色体,并且,考虑到STR位点仅能代表其所在染色体的部分区域,也会存在部分染色体结构异常和非整倍体无法通过STR区分的情况,再加上STR基因分型仅通过其位点峰图的相对高低来判断倍型,无法对胚胎的染色体倍型进行准确定量分析,根据STR位点判断染色体倍型也会有一定的局限性,对于胚胎的嵌合现象也不能进行准确的分析。此外,应用于PGT流程中的胚胎DNA是由3~5个单细胞的DNA经过MALBAC扩增后的产物,在进行STR分析过程中,部分STR位点可能会由于扩增造成检测过程中位点的不准确,本研究中患者RHM2的胚胎F1和F2也在STR基因分型中出现了杂峰信号,在实际应用过程中需要对峰图进行仔细核对。
辅助生育治疗对于规避HM的再发风险和癌变可能十分重要,植入前胚胎遗传学检测可降低HM的发生风险,保护女性患者的生育力。然而,行辅助生殖治疗的RHM患者临床样本较珍贵,不便用于科研。此外,由于HM病因复杂,既往有关PGT治疗在RHM患者中的应用没有针对性。而本研究利用临床上废弃的胚胎进行检测,极大程度的利用了临床剩余样本,同时本研究中入组的病人为不携带NLRP7/KHDC3L致病基因变异的非BiHM型RHM患者,此类患者的HM发生多为多精入卵或者是母源性遗传物质缺失导致的常见类型CHM/PHM,筛查胚胎的亲本来源和染色体倍型对于此类HM患者有很大的意义。本研究中基于NGS技术的PGT治疗在RHM患者中的应用提供了先验证据,最新的检测技术与创新的分析方法可以降低RHM发生风险,同时提高了RHM患者的妊娠率,但是本研究中技术方法的局限性主要在于通过筛选胚胎染色体的亲本来源和倍型异常仅仅适用于CHM或者PHM的患者,而不适用于BiHM型RHM患者。
本研究为应用于非BiHM型RHM患者胚胎植入前遗传学检测进行了探究,明确了基于NGS技术的PGT治疗方案通过杂合SNP数量和CNV对胚胎进行亲本来源和染色体倍型分析的可行性,为RHM患者的助孕治疗提供了技术支持。此外,本研究的阳性结果也为该方法应用于RHM患者的PGT进程中提供了新的思路。
1 计鸣良,赵峻,向阳.葡萄胎诊治的研究进展.中国癌症防治杂志,2020,12:126-130.
2 Moein-Vaziri N,Fallahi J,Namavar-Jahromi B,et al.Clinical and genetic-epigenetic aspects of recurrent hydatidiform mole:A review of literature.Taiwan J Obstet Gyne,2018,57:1-6.
3 Ning F,Hou H,Morse AN,et al.Understanding and management of gestational trophoblastic disease.F1000Res,2019,8.
4 Qian JH,Cheng Q,Murdoch S,et al.The genetics of recurrent hydatidiform moles in China:correlations between NLRP7 mutations,molar genotypes and reproductive outcomes.Mol Hum Reprod,2011,17:612-619.
5 Kalogiannidis I,Kalinderi K,Kalinderis M,et al.Recurrent complete hydatidiform mole:where we are,is there a safe gestational horizon? Opinion and mini-review.J Assist Reprod Genet,2018,35:967-973.
6 贾继晶.葡萄胎患者的临床疗效分析.心理月刊,2019,14:162.
7 Reubinoff BE,Lewin A,Verner M,et al.Intracytoplasmic sperm injection combined with preimplantation genetic diagnosis for the prevention of recurrent gestational trophoblastic disease.Hum Reprod,1997,12:805-808.
8 Hafezi M,Chekini Z ,Zamanian M.Which One Is More Prominent in Recurrent Hydatidiform Mole,Ovum or Sperm? Int J Fertil Steril,2020,14:154-158.
9 Ogilvie CM,Renwick PJ,Khalaf Y,et al.First use of preimplantation genotyping in prevention of recurrent diandric complete hydatidiform mole.Reprod Biomed Online,2009,19:224-227.
10 Sills ES,Obregon-Tito AJ,Gao H,et al.Pathogenic variant in NLRP7 (19q13.42) associated with recurrent gestational trophoblastic disease:Data from early embryo development observed during in vitro fertilization.Clin Exp Reprod Med,2017,44:40-46.
11 Baruch E ,Weller JI.Estimation of the number of SNP genetic markers required for parentage verification.Anim Genet,2008,39:474-479.
12 Bras A,Rodrigues AS ,Rueff J.Copy number variations and constitutional chromothripsis (Review).Biomed Rep,2020,13:11.
13 Wyner N,Barash M ,McNevin D.Forensic Autosomal Short Tandem Repeats and Their Potential Association With Phenotype.Front Genet,2020,11.