胚胎鼻骨于孕6周时开始发育,第9~11周时开始骨化[1]。产前超声筛查诊断胎儿鼻骨发育异常包括鼻骨缺失和鼻骨发育不良,被认为是胎儿非整倍体染色体异常的超声软指标之一,尤其是21三体[2-3]。但目前关于孤立性的鼻骨缺失或发育不良是否行介入性产前诊断意见不一,有研究认为孤立性的鼻骨缺失或发育不良染色体异常风险高,应行产前诊断[4-5],也有研究认为不增加胎儿染色体异常风险[6]。随着产前超声筛查技术的不断提升,越来越多的鼻骨发育异常胎儿被发现,关于是否合并染色体异常的报道较少,本文对166例鼻骨缺失或发育不良的胎儿进行回顾性研究,分析染色体核型与染色体微阵列分析(chromosomal microarray analysis,CMA)在鼻骨缺失或发育不良胎儿产前诊断中的应用,探讨产前筛查发现胎儿鼻骨发育异常是否有必要进一步行介入性产前诊断。
资料与方法
一、一般资料
收集2018年1月—2020年5月份在本院产检或外院转诊至本院,经产前超声筛查发现胎儿鼻骨缺失或鼻骨发育不良的胎儿资料进行回顾性分析。孕20~26周超声检查胎儿面部正中矢状面、横切面和冠状面均未显示鼻骨声像者诊断为鼻骨缺失;鼻骨超声测量值<2.5 mm诊断为鼻骨发育不良[7]。其中单纯性的鼻骨缺失或发育不良(孤立性组)共137例,合并其他微小异常或者结构异常(非孤立性组)共29例。孕妇年龄16~41岁,高龄孕妇11例,平均(28.5±5.1)岁,孕周18~25周。
二、方法
1.羊水标本采集:胎儿诊断为鼻骨缺失或发育不良的孕妇均进行优生遗传咨询,签署介入性产前诊断知情同意书,按无菌操作要求,经超声引导下行羊膜腔穿刺术,抽取羊水标本共30 mL,行染色体核型及CMA。
2.染色体G显带制备:羊水离心、去上清液留沉淀,接种于培养瓶中,经收获、固定、制片、60~80 ℃烘烤后行G显带;计数20个分裂相,分析5个核型,嵌合型计数增加至 50~100 个分裂相,依据《人类遗传学国际命名体制 ISCN(2016)》进行染色体核型命名。
3.CMA:采用Affymetrix Cytoscan 750 K(美国Affymetrix公司)芯片(约75万个探针位点)进行扫描检测,按照试剂盒步骤,样本基因组经酶切、消化、PCR扩增、纯化、片段化、标记、与芯片杂交、洗涤、扫描以及数据分析,应用ChAS软件进行数据分析,结果查询 OMIM、UCSC、DECIPHER、ISCA、DGV等数据库。如胎儿CMA异常,建议胎儿父母CMA比对,确定变异来源。
4.统计学方法:采用 SPSS 23.0统计软件,计量资料采用![]() 描述,计数资料采用频数及率描述,组间差异比较采用χ2检验。以P<0.05 为差异有统计学意义。
描述,计数资料采用频数及率描述,组间差异比较采用χ2检验。以P<0.05 为差异有统计学意义。
结 果
一、鼻骨缺失或发育不良胎儿的染色体检测结果
166例鼻骨缺失或鼻骨发育异常的胎儿羊水样本中,检测出染色体核型异常16例,检出率9.6%(16/166),染色体微阵列分析(CMA)异常19例,检出率11.5%(19/166),两者均异常共15例。共有22例鼻骨缺失或发育不良胎儿的染色体存在异常,见表1。
二、孤立性组与非孤立性组染色体G显带结果分析
孤立性鼻骨发育异常137例,其中染色体核型异常6例,检出率4.4%(6/137),均为21三体或21三体嵌合;非孤立性的鼻骨发育异常29例,其中染色体核型异常10例,检出率34.5%(10/29),8例为21三体,1例为47,XXX,1例为18p11(del),两组检出率比较差异有统计学意义。详见表1、表2。
表1 鼻骨发育异常胎儿的染色体异常情况
Table 1 Chromosomal abnormalities in fetuses with nasal bone dysplasia
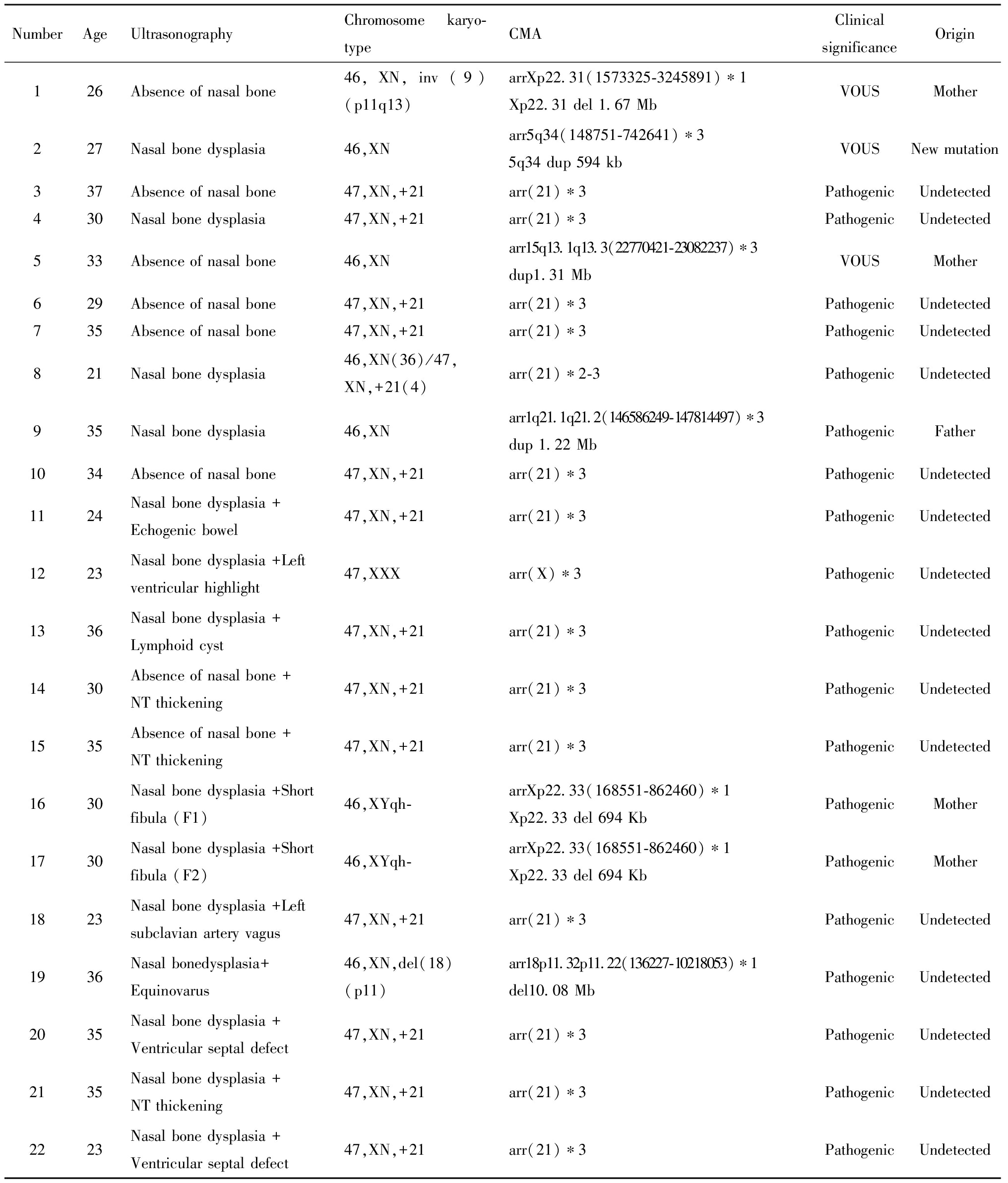
NumberAgeUltrasonographyChromosome karyo-typeCMAClinical significanceOrigin126Absence of nasal bone46,XN,inv(9)(p11q13)arrXp22.31(1573325-3245891)∗1 Xp22.31 del 1.67 MbVOUSMother227Nasal bone dysplasia46,XNarr5q34(148751-742641)∗35q34 dup 594 kbVOUSNew mutation337Absence of nasal bone47,XN,+21arr(21)∗3PathogenicUndetected430Nasal bone dysplasia47,XN,+21arr(21)∗3PathogenicUndetected533Absence of nasal bone46,XNarr15q13.1q13.3(22770421-23082237)∗3dup1.31 MbVOUSMother629Absence of nasal bone47,XN,+21arr(21)∗3PathogenicUndetected735Absence of nasal bone47,XN,+21arr(21)∗3PathogenicUndetected821Nasal bone dysplasia46,XN(36)/47,XN,+21(4)arr(21)∗2-3PathogenicUndetected935Nasal bone dysplasia46,XNarr1q21.1q21.2(146586249-147814497)∗3dup 1.22 MbPathogenicFather1034Absence of nasal bone47,XN,+21arr(21)∗3PathogenicUndetected1124Nasal bone dysplasia +Echogenic bowel47,XN,+21arr(21)∗3PathogenicUndetected1223Nasal bone dysplasia +Left ventricular highlight47,XXXarr(X)∗3PathogenicUndetected1336Nasal bone dysplasia +Lymphoid cyst47,XN,+21arr(21)∗3PathogenicUndetected1430Absence of nasal bone +NT thickening47,XN,+21arr(21)∗3PathogenicUndetected1535Absence of nasal bone +NT thickening47,XN,+21arr(21)∗3PathogenicUndetected1630Nasal bone dysplasia +Short fibula (F1) 46,XYqh-arrXp22.33(168551-862460)∗1Xp22.33 del 694 KbPathogenicMother1730Nasal bone dysplasia +Short fibula (F2)46,XYqh-arrXp22.33(168551-862460)∗1Xp22.33 del 694 KbPathogenicMother1823Nasal bone dysplasia +Left subclavian artery vagus47,XN,+21arr(21)∗3PathogenicUndetected1936Nasal bonedysplasia+Equinovarus46,XN,del(18)(p11)arr18p11.32p11.22(136227-10218053)∗1del10.08 MbPathogenicUndetected2035Nasal bone dysplasia +Ventricular septal defect47,XN,+21arr(21)∗3PathogenicUndetected2135Nasal bone dysplasia +NT thickening47,XN,+21arr(21)∗3PathogenicUndetected2223Nasal bone dysplasia +Ventricular septal defect47,XN,+21arr(21)∗3PathogenicUndetected
VOUS:variant of unknown significance; N.16 and N.17 are twins
表2 两组胎儿染色体G显带结果情况
Table 2 G-banding results of fetal chromosomes in the two groups
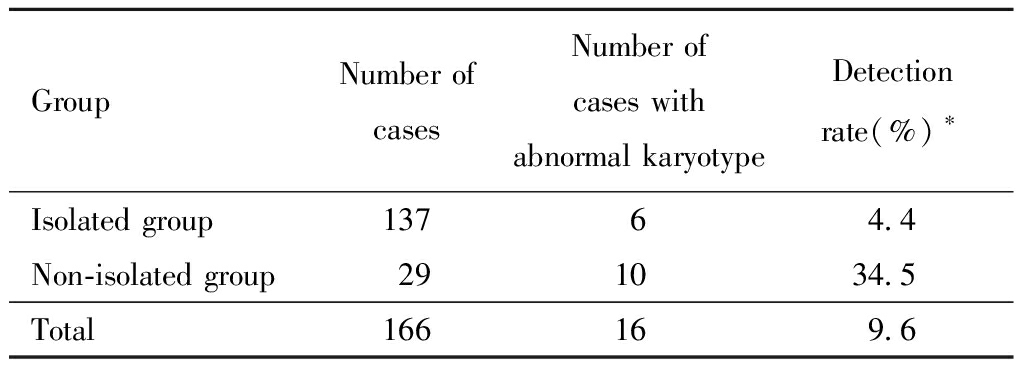
GroupNumber of casesNumber of cases with abnormal karyotypeDetection rate(%)∗Isolated group13764.4Non-isolated group291034.5Total166169.6
Notes:Comparison between the two groups,*P<0.005
三、孤立性组与非孤立性组CMA异常结果分析
孤立性组CMA检出致病性拷贝数变异(CNVs)7例,检出率5.1%(7/137),5例为21三体,1例为21三体嵌合,1例为1q21.1q21.2重复,父母行CMA比对,来源于父亲。另有CNVs临床意义未明(VOUS)3例,胎儿父母均行CMA比对,其中2例胎儿来源于母亲,1例父母未携带其胎儿检出CNVs。非孤立性组CMA检出致病性CNVs 12例,检出率41.3%(12/29),8例为21三体,1例为47,XXX,1例为18p11.32p11.22缺失,2例(双胎)为Xp22.33缺失,胎儿父母行CMA比对,来源于母亲,两组检出率比较差异有统计学意义。详见表1、表3。
表3 两组胎儿染色体微阵列分析(CMA)结果情况
Table 3 Results of fetal CMA in the two groups

GroupNumber of casesNumber of CMAexceptionsDetection rate(%)∗Isolated group13775.1Non-isolated group291241.3Total1661911.4
Notes:Comparison between the two groups,*P<0.005
讨 论
2001年Cicero等[8]首次报道了21三体综合征与胎儿鼻骨缺失有关。杨昕等[9]研究显示,孤立性鼻骨缺失或发育不良胎儿染色体异常发生率为12.7%,当合并其他结构异常时,染色体异常发生率上升至44.4%。本研究显示孤立性鼻骨发育不良染色体核型异常检出率4.4%,均为21三体综合征或21三体嵌合,其中高龄占比为2/5,CMA检测致病性CNVs的检出率为5.1%,其中21三体占比为6/7;鼻骨发育异常合并其他微小异常或结构异常时染色体异常明显上升,染色体核型检出率为34.5%,其中21三体占比为8/10,CMA检出率为41.3%,其中21三体占比为8/12。在本研究中胎儿鼻骨超声异常主要与21三体相关,与研究报道一致。本研究提示孤立性的胎儿鼻骨发育产前超声筛查异常,其染色体核型或CMA异常合计发生率高达7.3%(10/137),应建议行介入性产前诊断;非孤立性的胎儿鼻骨发育产前超声筛查异常,染色体核型及CMA异常发生率更高,强烈建议介入性产前诊断。
除21三体外,有少量的研究报道鼻骨缺如或鼻骨发育不良与性染色体、18三体有关[10-12]。本研究发现在胎儿染色体异常中,有1例性染色体增多为47,XXX,胎儿父母行外周血染色体正常,电话随访该胎儿引产;1例为18号染色体p11.32p11.22缺失10.08 Mb,为18号部分单体,因缺失片段较大,可能会出现染色体病的临床常见表型如智力障碍、生长发育迟缓、异常面容等,电话随访该胎儿引产。在胎儿染色体G显带正常下,CMA发现致病性CNVs 3例,1例为1q21.1q21.2 1.22 Mb重复,该重复表现为先天性心脏缺陷,发育迟缓,精神分裂征,外显率29.1%,该胎儿超声表现为单纯性的鼻骨发育不良,胎儿父母行CMA比对,该胎儿的致病性CNVs 来源于父亲,父亲表型正常,经考虑后选择继续妊娠;2例为双胎(IVF、双绒双羊)为Xp22.33 694 Kb缺失,该缺失表现为不成比例的肢体短小及身材矮小,前壁内旋等,该双胎超声表现为鼻骨发育不良、腓骨短,孕妇本人身高1.4 m,腓骨、肱骨短,智力正常,经CMA比对孕妇本人亦为Xp22.33 缺失,考虑变异来源于母亲,因孕妇本人卵巢功能低下,本次妊娠为辅助生殖助孕,选择继续妊娠。另外CMA发现3例临床意义未明CNVs,与DGV数据库重叠,正常人群和患病人群均可检出,经父母CMA比对,其中2例来源于母亲,1例为新发,均选择继续妊娠,该新发意义未明CNVs胎儿足月顺产,目前表观及智力方面未见异常。因此,本研究提示胎儿鼻骨发育异常行介入性产前诊断,需行染色体核型外,还需行染色体微阵列分析(CMA),CMA检测可排除某些微小基因片段缺失或重复导致的综合征可能[13-14],研究[15-16]表明CMA在胎儿超声异常可额外检出6%~7%的染色体异常,CMA的检出率达6%~27.4%[17-20],本文染色体核型检出率9.6%,CMA检出率16.1%,提高了6.5%,与文献报道类似。当胎儿CMA检出临床意义未明的CNVs时,应建议胎儿父母行CMA比对,明确变异来源,有助于遗传咨询。
综上所述,胎儿鼻骨超声异常应建议行介入性产前诊断,尤其是合并其他微小异常或结构异常,或年龄高风险,应强烈建议介入性产前诊断。若染色体核型正常,需行染色体微阵列分析(CMA),如胎儿CMA结果为临床意义未明,建议父母行CMA比对,有助于判断变异来源及进一步遗传咨询。
1 王慧芳,王诗雅.鼻骨发育异常筛查胎儿染色体异常的临床价值.中国产前诊断杂志(电子版),2015,7:8-10.
2 Gautier M,Gueneret M,Plavonil C,et al.Normal Range of Fetal Nasal Bone Length during the Second Trimester in an Afro-Caribbean population and likelihood ratio for trisomy 21 of absent or hypoplastic nasal bone.Fetal Diagn Ther,2017,42:130-136.
3 Cicero S,Rembouskos G,Vandecruys H,et al.Likelihood ratio for trisomy 21 in fetuses with absent nasal bone at the 11-14-week scan.Ultrasound Obstet Gynecol,2004,23:218-223.
4 Bethune M.Literature review and suggested protocol for managing ultrasound soft markers for Down syndrome:thickened nuchal fold,echogenic bowel,shortened femur,shortened humerus,pyelectasis and absent or hypoplastic nasal bone.Australas Radiol,2007,51:218-225.
5 马涛,杨晓,岳军,等.妊娠中晚期超声软指标与胎儿染色体异常及其围生结局.实用妇产科杂志,2017,33:110-113.
6 Ting YH,Lao TT,Lau TK,et al.Isolated absent or hypoplastic nasal bone in the second trimester fetus:is amniocentesis necessary.J Matern Fetal Neonatal Med,2011,24:555-558.
7 钱晓婷,包凌云,黄安茜,等.孕中晚期胎儿鼻骨的超声评估在染色体异常诊断中的价值.温州医科大学学报,2019,49:507-511.
8 Cicero S,Curcio P,Papageorghiou A,et al.Absence of nasal bone in fetuses with trisomy 21 at 11-14 weeks of gestation:an observational study.Lancet,2001,358:1665-1667.
9 杨昕,韩瑾,甄理,等.胎儿鼻骨缺失或发育不良与染色体核型异常的关系——187例分析.中华围产医学杂志,2015,18:339-342.
10 Agathokleous M,Chaveeva P,Poon LC,et al.Meta-analysis of second-trimester markers for trisomy 21.Ultrasound Obstet Gynecol,2013,41:247-261.
11 Lin CY,Wang PH,Yang MJ,et al.A case of 45,X/47,XYY mosaicism in a male fetus with a hypoplastic nasal bone.J Ultrasound Med,2015,34:353-354.
12 Kagan KO,Sonek J,Berg X,et al.Facial markers in second-and third-trimester fetuses with trisomy 18 or 13,triploidy or Turner syndrome.Ultrasound Obstet Gynecol,2015,46:60-65.
13 Dukhovny S,Wilkins-Haug L,Shipp TD,et al.Absent fetal nasal bone:what does it mean for the euploid fetus.J Ultrasound Med,2013,32:2131-2134.
14 刘丛丛,戚庆炜,蒋宇林,等.26例胎儿鼻骨发育异常的产前诊断结果分析.发育医学电子杂志,2020,8:34-41.
15 Levy B,Wapner R.Prenatal diagnosis by chromosomal microarray analysis.Fertil Steril,2018,109:201-212.
16 Ganapathi M,Nahum O,Levy B.Prenatal diagnosis using chromosomal SNP microarrays.Methods Mol Biol,2019,1885:187-205.
17 Lee CN,Lin SY,Lin CH,et al.Clinical utility of array comparative genomic hybridisation for prenatal diagnosis:a cohort study of 3171 pregnancies.BJOG,2012,119:614-625.
18 Shaffer LG,Dabell MP,Fisher AJ,et al.Experience with microarray-based comparative genomic hybridization for prenatal diagnosis in over 5000 pregnancies.Prenat Diagn,2012,32:976-985.
19 Hillman SC,Pretlove S,Coomarasamy A,et al.Additional information from array comparative genomic hybridization technology over conventional karyotyping in prenatal diagnosis:a systematic review and meta-analysis.Ultrasound Obstet Gynecol,2011,37:6-14.
20 Breman A,Pursley AN,Hixson P,et al.Prenatal chromosomal microarray analysis in a diagnostic laboratory; experience with >1000 cases and review of the literature.Prenat Diagn,2012,32:351-361.